En un estudio con conejos, científicos vieron que la toxina Shiga de Escherichia coli enterohemorrágica humana afecta dramáticamente al sistema inmunitario de las células epiteliales del intestino. Aquí se explica cómo se realizó dicho estudio y cómo se ven afectadas dichas células por esa enfermedad.
Presentación
“Shiga toxin remodels the intestinal epithelial transcriptional response to Enterohemorrhagic Escherichia coli” (“La toxina Shiga remodela la respuesta transcripcional del epitelio intestinal frente a Escherichia coli enterohemorrágica”) es un artículo científico elaborado por Alyson R. Warr, Carole J. Kuehl, y Matthew K. Waldor, y publicado en la revista PLOS Pathogens en febrero de 2021, en el cual sus autores, mediante un estudio con conejos infantiles, descubrieron que la toxina Shiga de Escherichia coli enterohemorrágica afecta de manera importante a la respuesta inmune de las células epiteliales del intestino.
Escherichia coli enterohemorrágica es un patógeno transmitido por los alimentos que causa enfermedades diarreicas y el síndrome urémico hemolítico potencialmente letal, y es responsable de hasta 2 millones de casos anuales de enfermedad gastrointestinal aguda. La infección está asociada con una respuesta inflamatoria en el colon, y los pacientes tienen niveles elevados de leucocitos fecales y calprotectina. Las muestras de biopsia colónica de pacientes con infección muestran inflamación, edema, depósito de fibrina, invasión de neutrófilos y hemorragia. La terapia de rehidratación de apoyo sigue siendo el tratamiento principal para la infección, ya que los antibióticos están contraindicados, además, no hay vacuna disponible.
Durante la infección de este patógeno, se libera una potente toxina en el intestino, la toxina Shiga, pero existe un conocimiento limitado de cómo esta toxina da forma a la respuesta del huésped a la infección in vivo, dado que los ratones no suelen manifestarla.
Por ello, los científicos y autores de este trabajo utilizaron un modelo de infección por este patógeno de conejo lactante, que imita de cerca la enfermedad humana, para evaluar las respuestas intestinales a la infección. Las comparaciones de la secuenciación de ARN específica del compartimento celular del tejido intestinal de cepas que contienen y carecen de toxina Shiga, revelaron que esta toxina remodela notablemente la forma en que el compartimento de las células epiteliales responde a la infección. En conjunto, estos hallazgos sugieren que la toxina Shiga de Escherichia coli enterohemorrágica da forma a la respuesta inmune innata intestinal a esta infección y proporciona información sobre el complejo diálogo huésped-patógeno que subyace a la enfermedad.

Métodos de trabajo
Aquí se narran los métodos llevados a cabo por los científicos en los experimentos sobre la toxina Shiga de Escherichia coli enterohemorrágica.
Uso de animales y de cepas bacterianas
Los experimentos con los conejos blancos de Nueva Zelanda se realizaron usando protocolos aprobados por el Comité de Animales del Brigham and Women’s Hospital y de acuerdo con las recomendaciones de la Guía del Instituto Nacional de Salud para el Cuidado y Uso de Animales de Laboratorio, y la Ley de Bienestar Animal del Departamento de Agricultura de los Estados Unidos.
Por su parte, todas las cepas bacterianas se cultivaron en medio LB o en placas de agar LB a 37°C. En todos los experimentos de este estudio se utilizó un mutante resistente a la gentamicina de E. coli O157:H7 cepa EDL933, construido mediante una recombinación de rojo lambda.
Infección de los conejos lactantes y procesamiento de tejidos
Después de preparar los inóculos de infección, cada conejo recién nacido se inoculó orogástricamente con 500μl de este, utilizando un catéter French de tamaño 4. Después de la inoculación, los conejos se monitorearon al menos 2 veces al día para detectar signos de enfermedad y se sacrificaron 2 días (36 a 40 horas) después de la infección, cuando se les extrajo todo el tracto intestinal.
De ese tracto intestinal, se extrajeron secciones de un cm del colon medio y distal después de la necropsia. Las series de dilución se sembraron en placas LB Gm que se incubaron, para determinar las cargas bacterianas de CFU/g en las secciones de tejido.
Conservación de tejidos e histopatología
Para la conservación de los tejidos, se fijaron secciones de dos cm de colon medial y distal en 2ml de formalina tamponada neutra al 10% durante la noche (~16 horas) a temperatura ambiente. Al día siguiente, las secciones de tejido se transfirieron a 2ml de etanol al 70%.
Para realizar el estudio de histopatología, secciones de 5μm incrustadas en parafina y fijadas en formalina, se tiñeron con hematoxilina y eosina (H&E) en el Instituto de cáncer Dana Farber, donde fueron evaluados a ciegas por un histopatólogo, y puntuados semicuantitativamente, según lo siguiente:
- Se evaluó la inflamación de las secciones utilizando los siguientes criterios: 0, ninguno; 1, infiltración leve de células inmunitarias en la lámina propia; 2, infiltración moderada; 3, infiltración extensa; 4, infiltración severa y extensa.
- La apoptosis se evaluó utilizando los siguientes criterios: 0, ninguno; 1, pocas células observadas con núcleos fragmentados; 2, muchas células con núcleos fragmentados; 3, núcleos apoptóticos significativos y penetración a las criptas; 4, apoptosis transmural.
- El edema, la congestión y la hemorragia se evaluaron utilizando los siguientes criterios: 0, ninguno; 1, congestión vascular leve y/o edema leve; 2, congestión y/o edema moderado; 3, congestión con hemorragia +/ edema; 4, congestión con hemorragia multifocal severa +/- edema.
- La infiltración de heterófilos se evaluó utilizando los siguientes criterios: 0, ninguno; 1, heterófilos individuales dispersos o pequeños grupos en la lámina propia; 2, agregados multifocales en la mucosa con pocas células en el lumen; 3, agregados multifocales en la mucosa con abundante extrusión de células en el lumen; 4, agregados multifocales en mucosa con grandes balsas heterófilas intraluminales.
- El desprendimiento se evaluó utilizando los siguientes criterios: 0, ninguno; 1, pocas células epiteliales desprendidas de la superficie luminal; 2, número moderado de células epiteliales desprendidas de la superficie luminal; 3, la superficie epitelial está gravemente alterada; 4, desprendimiento extenso y severo (falta la capa epitelial).
Después, las puntuaciones se compararon los tipos de infección utilizando una prueba estadística Mann-Whitney U de dos colas.

Preparación de tejidos para secuenciación de ARN
Con el fin de llevar a cabo una secuenciación de ARN, se recolectaron secciones de cinco cm entre el colon medio y distal de 3 conejos inoculados. Se aislaron fracciones de células epiteliales y de células de la lámina propia, las cuales se almacenaron a -80°C hasta la extracción del ARN.
Después de la disociación de las células epiteliales, la pieza de tejido restante se transfirió a un tubo que contenía 5ml de solución de digestión enzimática. Se detuvo la digestión y se filtró la solución hasta un volumen final de 30ml, el cual se centrifugó y el sedimento celular se resuspendió en 2ml de Trizol. Finalmente, estas muestras también se almacenaron a -80°C hasta la extracción del ARN.
Extracción de ARN y preparación y secuenciación de bibliotecas de secuencias de ARNm
El ARN se extrajo del Trizol (una vez descongeladas las muestras), utilizando el kit Direct-Zol RNA MiniPrep Plus de Zymo con algunas modificaciones.
Se evaluó la calidad e integridad del ARN total (RINe) utilizando una cinta de detección de ARN de alta sensibilidad (Agilent) en las instalaciones de biopolímeros de HMS.
Se preparó ARN de alta calidad para la secuenciación de ARNm usando el kit KAPA mRNA HyperPrep (Roche).
Las bibliotecas de ARNm se cuantificaron utilizando una cinta de pantalla D1000 de alta sensibilidad (Agilent) y un Qubit de alta sensibilidad. Las bibliotecas se secuenciaron en un NextSeq 550.

Análisis genómicos, enriquecimiento de conjuntos de genes y comparación de genes expresados en tejidos infectado y no infectados
Para estudiar la expresión de los genes, los científicos crearon un archivo FASTA del genoma de conejo de referencia que se cargó en la plataforma web Galaxy junto a las lecturas de secuenciación para ser procesado y mapeado.
Las matrices de recuento se exportaron desde Galaxy y se importaron al programa R (versión 3.5.3), donde la abundancia de transcritos de genes marcadores se comparó usando una prueba no paramétrica U de Mann-Whitney de dos colas.
La matriz de recuento no normalizada también se analizó para comparar la abundancia de transcripciones entre diferentes tipos de inóculo, para identificar genes expresados diferencialmente.
Por otro lado, los genes se ordenaron por rango (calculado multiplicando el signo del cambio de pliegue por el valor p ajustado transformado log10), mientras que el agrupamiento jerárquico de muestras, se realizó con Heatmap.2 construyendo un dendrograma por columna (muestras). Los mapas de calor se escalaron por fila y los colores se asignaron utilizando la paleta RdYlBu de ColorBrewer (versión 1.1.2).
El enriquecimiento del conjunto de genes se realizó utilizando GSEA rápido (fGSEA) en R (versión 1.8.0). Solo se consideraron los genes con anotación. Se utilizaron los conjuntos de genes distintivos y los conjuntos de genes de firmas inmunológicas de MSigDB. Se completaron 1000 permutaciones y se excluyeron las categorías con menos de 10 genes y más de 500 genes. Los genes con un valor de p ajustado de <0,05 y un cambio de veces log2 de >2 o <-2 de DESeq2, se consideraron expresados diferencialmente.

Hibridación de ARN fluorescente in situ
La hibridación fluorescente in situ (FISH) se realizó mediante el ensayo RNAscope (ACDBio) Multiplex Fluorescent V2 en 5 secciones diferentes de tejido de colon de conejo por tipo de inóculo. Se siguieron los protocolos del fabricante y se utilizó el horno RNAscope HybEZ para todas las incubaciones.
Después de completar el ensayo de RNAscope, las secciones se procesaron más para la inmunofluorescencia.
Análisis cuantitativo de imágenes
Se tomaron imágenes de las muestras con un microscopio Nikon Ti Eclipse equipado con una cámara de campo amplio Andor NeoZyla y un objetivo de 20x.
Para las secciones teñidas con H&E, se realizó la alineación de Kohler y el balance de blancos, y luego se capturaron las imágenes RGB.
Para las secciones teñidas con anticuerpos fluorescentes y sondas FISH, se capturaron imágenes unidas de 4×6 (10% de superposición) para cada sección de tejido, mediante adquisición multicanal (azul, rojo, rojo lejano) e imágenes de 16 bits.
En ImageJ/FIJI se establecieron los valores de umbral para cada canal. Emplearon la herramienta FIJI «crear selección» para dibujar una selección, la cual se transfirió al canal de RNAscope y se registró el área de la señal de RNAscope dentro de esta área. Así, calcularon el área de tejido que expresa la señal, el porcentaje de la señal de RNAscope derivada de las células epiteliales y el porcentaje de señal dentro de las células epiteliales. Estas tres cuantificaciones se compararon para cada tipo de infección para cada sonda, mediante una prueba estadística Mann-Whitney U de dos colas.

Infección en cultivo tisular y RT-qPCR
Para realizar un estudio de la infección en cultivo, los científicos compraron células de adenocarcinoma colorrectal de colon humano y las cultivaron. Dos días antes de la infección de estas, sembraron 500.000 células en placas de 6 pocillos, de modo que las infecciones se produjeran con una confluencia de aproximadamente el 75%. Para ello, añadieron 45μl de cada inóculo bacteriano a 5 de los 6 pocillos.
Pasadas 6 horas de la infección, extrajeron el ARN con el kit RNeasy Plus Mini (Qiagen) y el ADNc se generó a partir de 2μg de ARN con un kit de transcripción inversa de ADNc de alta capacidad (Thermo Fisher), y se realizó una PCR cuantitativa en tiempo real con una máquina de PCR en tiempo real Step One Plus.
Finalmente, los niveles de expresión se compararon utilizando una prueba t de Student de dos colas.

Resultados del estudio sobre cómo afecta la toxina Shiga de Escherichia coli enterohemorrágica
A continuación, se indican los resultados obtenidos por los científicos en cada experimento de este estudio sobre la toxina Shiga de Escherichia coli enterohemorrágica.
La toxina Shiga de Escherichia coli enterohemorrágica promueve la apoptosis y la hemorragia en la mucosa colónica
Para investigar cómo la toxina Shiga de Escherichia coli enterohemorrágica modifica la respuesta de la mucosa colónica a la infección, los científicos utilizaron un aislado clínico (WT) prototípico de E. coli, de la que también obtuvieron la cepa mutante ΔΔstx. Ambas, WT y ΔΔstx fueron administradas a los conejos lactantes por vía orogástrica.
Tras esto, vieron que la toxina Shiga no altera la colonización de E. coli enterohemorrágica, pero sí que contribuye al desarrollo de diarrea (89% de diarrea en animales inoculados con WT frente a 40% en animales inoculados con ΔΔstx).
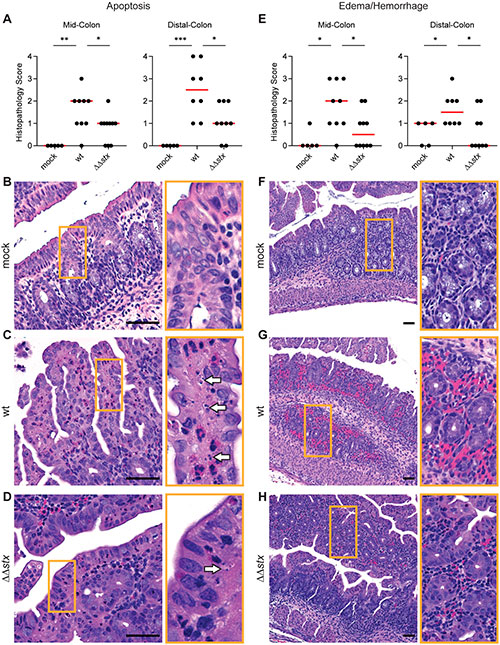
Por su parte, los análisis histopatológicos de muestras de colon de animales inoculados con WT, ΔΔstx o PBS (simulado) se llevaron a cabo en el pico de colonización (36 a 40 horas después de la inoculación). En comparación con los animales de control tratados de forma simulada, las muestras de colon de conejos infectados con WT y ΔΔstx tenían cambios patológicos prominentes en el colon medio y distal, sitios de máxima colonización.
Las infecciones por WT y ΔΔstx condujeron a niveles similares de inflamación general del colon, caracterizada por un mayor número de células mononucleares pequeñas en el tejido submucoso, así como niveles comparables de infiltración de heterófilos (neutrófilos lapinos). Tanto la infección por WT como por ΔΔstx provocaron un desprendimiento epitelial menor en el colon. Sin embargo, en comparación con la patología colónica asociada con la cepa ΔΔstx, hubo una apoptosis significativamente mayor, indicada por núcleos fragmentados generalizados; y edema/hemorragia, indicada por acumulación generalizada de sangre y líquido en el tejido, en muestras de animales infectados con WT, tal y como se muestra en la siguiente figura, donde las flechas blancas de las imágenes señalan los núcleos apoptóticos.
La toxina Shiga da forma a la respuesta transcriptómica de la mucosa del colon
Para investigar más a fondo cómo la toxina Shiga de Escherichia coli enterohemorrágica modifica la respuesta de la mucosa colónica a la infección, utilizaron RNA-seq para caracterizar los transcriptomas de las células epiteliales colónicas y de la lámina propia derivadas de conejos lactantes inoculados orogástricamente con WT, ΔΔstx o PBS (simulacro).
En el colon, las células epiteliales reciben el contacto inicial con la bacteria y la toxina. Debajo de la capa epitelial, las células de la lámina propia, incluidas las células estromales e inmunitarias, responden a las señales de las células epiteliales o potencialmente al contacto directo con los PAMP para desencadenar respuestas inmunitarias adicionales.
Para perfilar los cambios transcripcionales en estas poblaciones celulares, recolectaron dos puntos en el momento de la colonización máxima y realizaron protocolos de enriquecimiento para células epiteliales y de lámina propia de 3 conejos por tipo de inóculo.
El ARN se extrajo de estas fracciones celulares, se sometió a secuenciación y se cartografió en el genoma del conejo. Se comparó la expresión normalizada de genes marcadores asociados con células epiteliales, estromales e inmunitarias entre fracciones epiteliales y de lámina propia. Cabe indicar que detectaron enriquecimiento en la Gb3-sintasa A4GALT, en la fracción de células epiteliales, validando datos previos de que el receptor de la toxina Shiga está presente en tejido de conejo infantil.
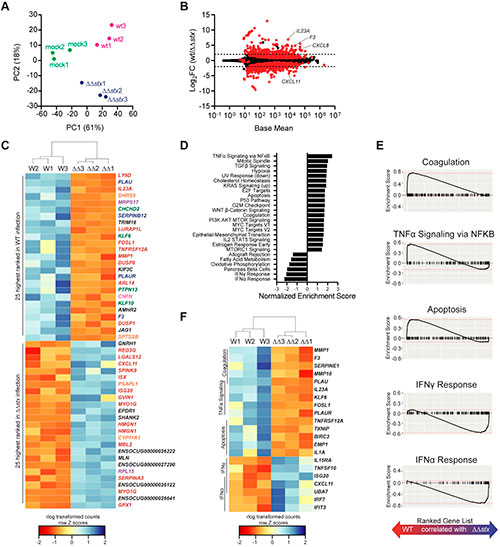
A continuación, los perfiles de expresión génica global para muestras individuales se evaluaron y compararon utilizando el paquete de expresión génica diferencial DESeq2. El análisis reveló que las muestras epiteliales de cada grupo, se agruparon por separado, destacando la especificidad de las respuestas transcriptómicas provocadas por cada tipo de inoculación en este compartimento. Sin embargo, las muestras de lámina propia no se segregaron de forma tan clara, lo que sugiere que la respuesta transcripcional a la toxina se concentra en el compartimento epitelial.
En relación con las muestras infectadas simuladas, la infección por WT estimuló más cambios transcriptómicos que la infección por ΔΔstx en las células epiteliales del colon. De los ~30 000 genes de conejo testeados, 1126 tenían una expresión significativamente diferente en WT/simulacro; mientras que para ΔΔstx/mock, se expresaron diferencialmente 497 genes. Por otro lado, 321 genes se expresaron diferencialmente de manera similar en ambos tipos de infección. La mayoría de estos genes compartidos se asociaron con los términos de función molecular GO comúnmente relacionados con la infección bacteriana, incluida la señalización y unión de citoquinas/quimioquinas, y el reconocimiento de LPS a través de la señalización de TLR4, lo que implica que muchas vías típicas de respuesta a patógenos se activan de manera similar en presencia o ausencia de la toxina. También se observaron patrones similares en el transcriptoma de la lámina propia.
Y aunque las cepas WT y ΔΔstx estimularon un conjunto de transcripciones compartidas, también encontraron diferencias sólidas y generalizadas en las respuestas transcriptómicas epiteliales a la infección por WT frente a ΔΔstx. Así, 390 genes exhibieron una expresión significativamente diferente entre estas dos condiciones, donde muchos de los 50 genes principales estaban involucrados en procesos relacionados con la coagulación y la señalización inmunitaria.
Se realizó un análisis de enriquecimiento de conjuntos de genes (GSEA) para identificar aún más los procesos transcripcionales asociados con la infección por WT o ΔΔstx. Con esto, identificaron una serie de vías asociadas específicamente a cada tipo de infección. En particular, la apoptosis, la coagulación y la señalización de NFκB, se asociaron con la respuesta transcriptómica a la infección por WT; mientras que las vías vinculadas con la señalización de IFNα e IFNγ, se asociaron con la infección por ΔΔstx. Asimismo, los impulsores de la coagulación, así como las citoquinas proinflamatorias IL23A e IL1A, se asociaron específicamente con la infección por WT, mientras que en la infección por ΔΔstx, muchos genes estimulados por interferón (ISG) se regularon diferencialmente. En conjunto, estos análisis, que pueden verse en la siguiente figura referida a los perfiles de las respuestas transcripcionales de las células epiteliales, sugieren que la toxina Shiga de Escherichia coli enterohemorrágica da forma a la respuesta inmune innata de las células epiteliales a esta infección.
Por otro lado, en muestras de la lámina propia, muchos menos genes exhibieron expresión diferencial en las comparaciones entre infecciones WT y ΔΔstx que en las muestras epiteliales (91 frente a 390). En esta ocasión, la agrupación jerárquica del transcriptoma completo no separó los perfiles transcripcionales de los colones infectados con WT y ΔΔstx tan claramente como se observaba en las muestras epiteliales; sin embargo, el análisis de agrupamiento mostró que los 50 genes principales por rango se distinguían por tipo de inóculo, muchos de los cuales funcionan en las vías de coagulación y señalización inmunitaria.
Tras realizar GSEA, se vio que las firmas asociadas con respuestas inmunitarias a patógenos extracelulares están presentes en la lámina propia. Los conjuntos de genes asociados con la infección por ΔΔstx incluyeron el «Virus de Newcastle», que induce una fuerte respuesta de interferón. El examen del subconjunto de vanguardia de estas vías subrayó que los genes asociados con las células Th17 están asociados con la infección WT, mientras que los genes estimulados por IFNγ, como CXCL11, están asociados con la infección ΔΔstx en la lámina propia, lo cual puede verse en la siguiente figura, relativa a los perfiles de las respuestas transcripcionales de las células de la lámina propia del colon. Juntos, estos análisis sugieren que en la lámina propia la toxina Shiga de Escherichia coli enterohemorrágica induce la expresión de genes típicamente asociados con células inmunes tipo 3 y que, en ausencia de esta toxina, las vías relacionadas con IFN son más prominentes.
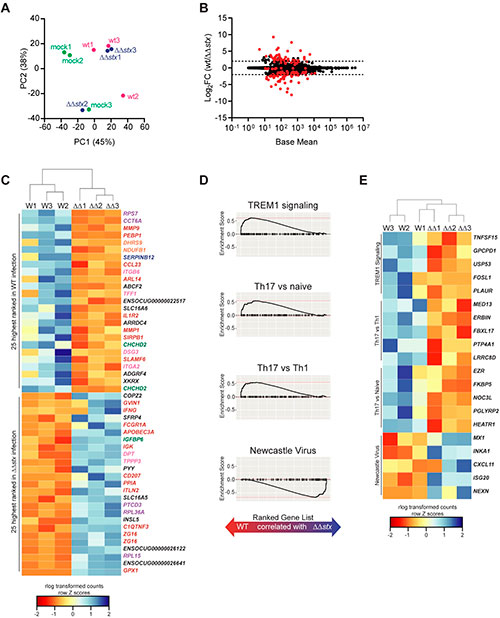
La infección de Escherichia coli enterohemorrágica estimula la expresión de genes asociados a la coagulación de manera dependiente de la toxina Shiga
Se sabe que la toxina Shiga de Escherichia coli enterohemorrágica causa trombosis en lechos vasculares fuera del tracto gastrointestinal y se ha investigado en la microvasculatura renal, pero, comparativamente, pocos estudios se han centrado en el intestino.
En este estudio, la comparación de los perfiles de expresión de muestras epiteliales y de lámina propia de animales infectados con WT frente a ΔΔstx, reveló diferencias en muchos genes asociados con la coagulación. Las muestras de infección por WT tenían niveles más altos de transcripciones para F3, un gen que codifica el iniciador de la cascada de la coagulación; las MMP y las SERPIN, que son proteasas que regulan el procesamiento de las proteínas de la cascada de la coagulación; así como la uroquinasa y el receptor de la uroquinasa, que regulan la fibrina.
La intensidad de fluorescencia media (MFI) del estudio de ARN FISH, fue mucho mayor en las muestras WT frente a ΔΔstx, y en las últimas muestras no difirió de los valores encontrados en las muestras infectadas simuladas, lo que sugiere que la toxina Shiga es fundamental para estimular la expresión de F3. En particular, casi toda (>90%) la señal de hibridación de F3 en muestras WT, se detectó en células que expresaban E-cadherina, un marcador epitelial. La uniformidad de la expresión de F3 a lo largo del epitelio, incluso en secciones donde se detectaron pocas células infectadas, sugiere que la toxina Shiga puede difundirse a lo largo del epitelio para modificar los programas transcripcionales en células que no tienen infección; por lo que se sugiere que las células epiteliales con infección pueden secretar factores que modifican la transcripción en las células vecinas.
Por otra parte, los autores del estudio también analizaron si la toxina Shiga estimula la transcripción de F3 en células HT29, una línea celular epitelial colónica humana. Después de llevar a cabo una qPCR tras una infección con WT, ΔΔstx EHEC o PBS (simulado), vieron que en la infección por WT, la expresión del gen F3 fue 5 veces mayor que en las células no infectadas; además, al igual que en los conejos, la inducción de la expresión de F3 en células HT29 dependía en gran medida de la toxina y hubo poca diferencia en la expresión de F3 en células no infectadas frente a aquellas infectadas con infección por ΔΔstx. Sin embargo, la capacidad del mutante ΔΔstx para inducir F3 en células HT29 se restableció mediante la adición de Stx2 puro, la cual también indujo la expresión de F3.
Por otro lado, se ha informado que la toxina Shiga conduce a aumentos en la actividad de la proteína procoagulante del factor tisular en las células del túbulo proximal renal y las células endoteliales, y se cree que este efecto se debe principalmente a una disminución en la expresión del inhibidor de la proteína del factor tisular (TFPI) y no a un aumento en la expresión del gen F3. Así que, también midieron los niveles de transcripción de TFPI en las mismas muestras utilizadas para medir la expresión de F3, para evaluar si esta vía también está activa en las células HT29. Tras lo cual, vieron que tanto la infección por WT como por ΔΔstx, estimularon de manera similar la expresión de transcritos de TFPI >10 veces, en comparación con las células no infectadas. Por lo tanto, la toxina Shiga parece regular el factor tisular por diferentes mecanismos en las células epiteliales del colon frente a las células endoteliales.
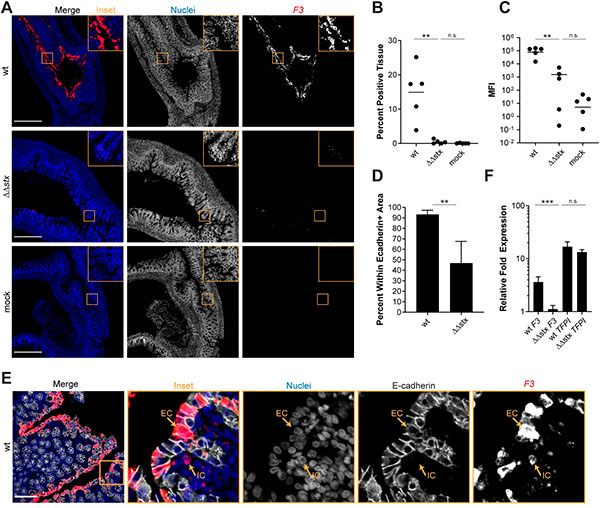
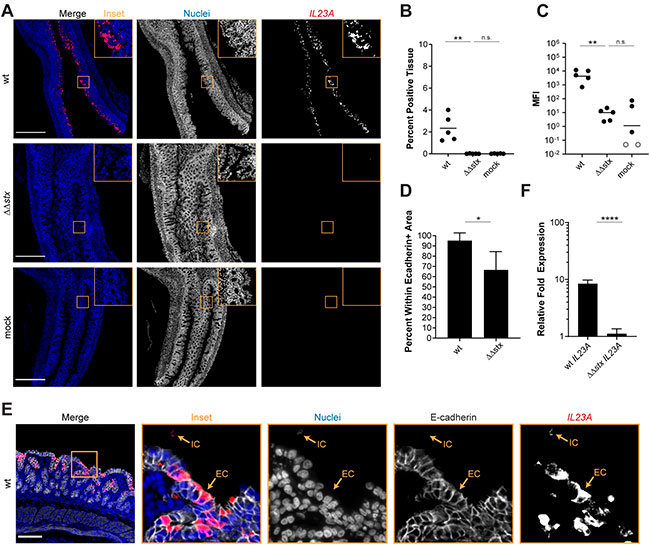
La toxina Shiga de Escherichia coli enterohemorrágica altera la expresión del gen de las citocinas
Los científicos emplearon RNA FISH para comparar la intensidad de fluorescencia y la distribución de las transcripciones de citocinas proinflamatorias IL23A en colones de conejos infectados con WT o ΔΔstx. La intensidad y el porcentaje de tejido que expresa la señal de IL23A en muestras de animales infectados con WT fueron sorprendentemente más altos que en animales infectados con ΔΔstx. Estos valores no difirieron en ΔΔstx y muestras de control, lo que sugiere que la toxina Shiga estimula la expresión de IL23A. Asimismo, inesperadamente, casi toda la expresión de IL23A en las muestras WT se detectó dentro de las células epiteliales (células positivas para E-cadherina), en comparación con las células de la lámina propia.
También validaron este hallazgo utilizando células HT29 cultivadas en tejido; donde se indujo la expresión de IL23A en células infectadas por WT, pero no por ΔΔstx. De manera similar a los hallazgos anteriores con la expresión de F3, la adición de Stx2 puro restauró la capacidad de ΔΔstx para estimular la expresión de IL23A a niveles de WT, por lo que Stx2 por sí mismo también induce IL23A.
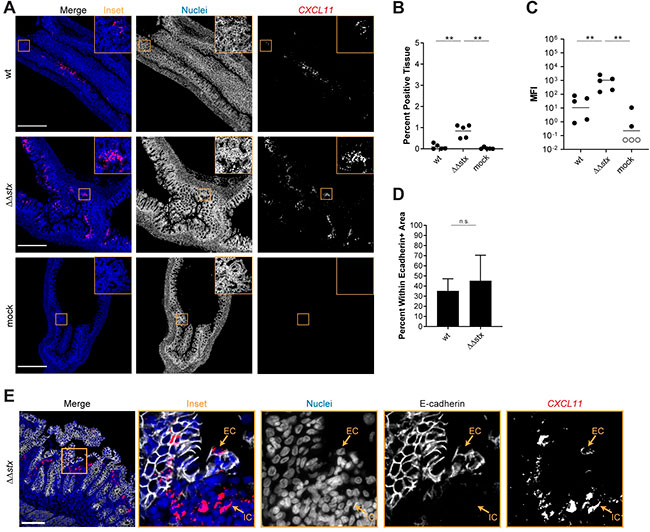
IL23 también promueve la expresión de otras citocinas, como la quimiocina CXCL8, que es un quimioatrayente de neutrófilos. Similar a la expresión de F3 e IL23A, las transcripciones de CXCL8 se observaron con una intensidad mucho mayor y en un área de tejido mucho más grande en muestras de infección por WT frente a ΔΔstx. Además, estas transcripciones de CXCL8 estaban presentes principalmente en las células epiteliales. También similares a F3 e IL23A, las señales de CXCL8 fueron uniformes en todo el tejido epitelial, sin mostrar una correlación aparente con los focos de infección a lo largo del epitelio. Juntas, estas observaciones sugieren que la toxina Shiga de Escherichia coli enterohemorrágica estimula la expresión de genes relacionados con citocinas en el epitelio del colon durante la infección.
Por otro lado, en los datos de RNAseq, encontraron que los animales infectados con ΔΔstx tenían niveles estadísticamente más altos de transcritos de IFNG que los infectados con WT, lo que sugiere que Shiga puede inhibir la producción de IFNγ, el cual es producido principalmente por células Th1, ILC1, células NK y macrófagos en la lámina propia.
A su vez, la señalización de IFNγ estimula la transcripción de ISG, muchos de los cuales se encontraron expresados diferencialmente en animales infectados con WT, frente a ΔΔstx. Uno de esos genes, es CXCL11, una quimiocina inducible por IFNγ producida por macrófagos que actúa como quimioatrayente para las células T CXCR3+, donde la participación de los macrófagos promueve el desarrollo de las células Th1. Los análisis de RNA FISH revelaron que la MFI de la señal CXCL11 fue más intensa y se detectó en más área de tejido en muestras de animales infectados con ΔΔstx frente a WT. Aproximadamente el 50% de la señal de transcripción de CXCL11 se encontró en células epiteliales y el resto en células E-cadherina negativas en la lámina propia. Estas observaciones sugieren que la toxina Shiga de Escherichia coli enterohemorrágica inhibe la señalización de IFNγ en el epitelio colónico durante la infección.
Discusión y Conclusión
La inoculación orogástrica de conejos lactantes con Escherichia coli enterohemorrágica conduce a una enfermedad diarreica y una patología del colon que se parece mucho a muchos de los aspectos intestinales de la misma infección humana, por ello, en este estudio se usó este modelo para estudiar cómo la toxina Shiga de Escherichia coli enterohemorrágica modifica la respuesta del huésped a la infección en el colon, comparando la histopatología y los perfiles transcripcionales de la mucosa colónica de animales infectados con WT o un mutante isogénico que carece de genes de la toxina (ΔΔstx).
Con esto, descubrieron que la toxina Shiga de Escherichia coli enterohemorrágica es una toxina potente que aumenta la apoptosis y la hemorragia en el tejido colónico y remodela drásticamente la respuesta transcripcional del epitelio colónico a la infección.
Aunque las respuestas transcripcionales provocadas por la infección por WT y ΔΔstx exhibieron cierta superposición, particularmente en las vías comúnmente asociadas con la infección por gramnegativos, estas cepas isogénicas provocaron perfiles transcripcionales muy distintos en el epitelio del colon. Casi el doble de genes se regularon diferencialmente en animales infectados con WT/simulado frente a ΔΔstx/simulado. Además, las diferencias en la respuesta epitelial atribuible a la toxina Shiga fueron particularmente prominentes en las vías de señalización y coagulación inmunitarias, lo que sugiere que esta toxina modifica fundamentalmente la respuesta inmunitaria innata intestinal a la infección por Escherichia coli enterohemorrágica.
Por otro lado, hay que indicar que la respuesta del huésped a esta infección ha sido difícil de estudiar directamente en humanos. El examen del suero y la orina de los pacientes ha revelado que la enfermedad grave se asocia con un aumento en la producción de citoquinas, incluidas CCL2, CCL4, CXCL1, CXCL5, CXCL8, CSF3, IL6, TNFα. La respuesta celular a la toxina Shiga se ha caracterizado más extensamente en células cultivadas en tejidos. La «respuesta de estrés ribotóxico» al daño celular mediado por esta toxina conduce a la regulación positiva de una variedad de genes y la producción de proteínas que modulan la respuesta inmune. Asimismo, la toxina Shiga purificada promueve la producción de factores de transcripción y citoquinas inflamatorias en cultivos células epiteliales e inmunitarias.
En este estudio descubrieron que varios de estos genes se expresaron de manera diferencial en colones infectados con WT frente a ΔΔstx; por lo que por sí misma, la toxina Shiga puede explicar un subconjunto de los cambios transcripcionales que identificaron en el epitelio del colon de animales infectados con la cepa WT.
Las comparaciones de los perfiles transcripcionales epiteliales inducidos por WT y ΔΔstx en colones de conejo sugieren que la toxina Shiga de Escherichia coli enterohemorrágica altera la expresión de citocinas importantes de formas imprevistas. La expresión de la citoquina IL23, fue notablemente mayor en los colones infectados con WT que con ΔΔstx. Se cree que IL23 estimula las respuestas inmunitarias de tipo 3 a los patógenos extracelulares, pero varios de estos hallazgos argumentan en contra de clasificar las respuestas que observaron como señalización inmunitaria de tipo 3. En primer lugar, generalmente se cree que la IL23 es producida por células dendríticas. Inesperadamente, casi toda la señal de IL23A se detectó en células epiteliales colónicas en colones de conejo. En segundo lugar, no detectaron transcripciones epiteliales de IL12B, la otra subunidad de la proteína IL23, cuando normalmente, las dos subunidades se expresan juntas, pero la expresión génica de estas dos subunidades no siempre está sincronizada temporalmente. Y, en tercer lugar, no detectaron transcripciones de IL17A, una citocina inducida por IL23, en la fracción celular de la lámina propia. Descifrar las funciones de IL23A en ausencia de IL12B puede revelar un aspecto no reconocido de la inmunidad de la mucosa epitelial; una posibilidad es que tenga una función independiente aparte, o también puede haber compañeros de unión alternativos para ella en las células epiteliales intestinales.
La toxina Shiga de Escherichia coli enterohemorrágica también parece inhibir la expresión de IFNG y muchos ISG como CXCL11, ya que estas citocinas y los factores en cascada estaban regulados a la baja en WT, en comparación con los colones infectados con ΔΔstx. Estudios in vitro anteriores también han demostrado que la toxina Shiga puede suprimir la señalización mediada por IFNγ y que el IFNγ circulante es bajo en pacientes humanos infectados. El IFNγ se expresa normalmente como parte de la respuesta inmunitaria de tipo 1, la falta de señalización de IFNG en colones infectados con Escherichia coli enterohemorrágica implica que este tipo de activación inmunitaria probablemente no ocurra en presencia de toxina Shiga.
El trabajo futuro debería cuantificar la abundancia de subtipos de células inmunitarias, como Th17, Th1 e ILC, durante la infección por Escherichia coli enterohemorrágica con y sin toxina Shiga. Sin embargo, actualmente no hay reactivos disponibles para el perfil inmunológico en conejos. La disponibilidad de reactivos mejorados para la identificación de células también permitirá perfeccionar los protocolos de enriquecimiento de células para futuros estudios transcriptómicos en poblaciones de células purificadas.
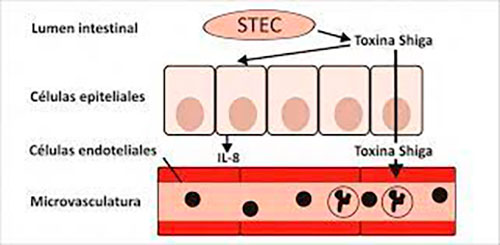
En el colon, las biopsias de pacientes han revelado que la infección por Escherichia coli enterohemorrágica también puede inducir trombos microvasculares y depósito de fibrina, pero pocos estudios han investigado los patrones de expresión génica que pueden contribuir a la trombosis en el intestino. Aunque los científicos de este estudio no pudieron detectar el depósito de fibrina en las secciones de tejido, vieron evidencia de daño vascular en forma de hemorragia y edema, los cuales fueron más prominentes en el tejido infectado con WT frente al infectado con ΔΔstx, aunque las muestras de ΔΔstx tenían una patología detectable. Por lo tanto, puede haber mecanismos tanto dependientes como independientes de la toxina Shiga que conduzcan a un aumento del edema y del daño vascular.
También encontraron que F3, el gen que codifica el iniciador de la cascada de la coagulación, se induce dramáticamente en las células epiteliales del colon en la infección por WT, pero no por ΔΔstx. De manera similar, las transcripciones de F3 se indujeron en HT29 (una línea celular de cáncer de colon humano) cultivado en tejido por WT, pero no por ΔΔstx. Stx2 en sí parece ser suficiente para estimular la expresión de F3 en las células epiteliales, ya que vieron que la adición de Stx2 exógena a células HT29 cultivadas en tejido, inducía la expresión de transcritos de F3 a niveles similares observados con la infección por WT. El factor tisular normalmente se expresa en células que no están en contacto con la sangre, como las células epiteliales y puede inducirse en respuesta a estímulos inflamatorios. En las células endoteliales, la toxina Shiga puede promover la actividad del factor tisular a través de un mecanismo asociado con una disminución en la expresión del inhibidor de la proteína del factor tisular (TFPI). Sin embargo, en células epiteliales colónicas de conejo y en células HT29, la expresión del gen TFPI no se alteró a pesar de un marcado aumento en la expresión del factor tisular, lo que sugiere un mecanismo alternativo de inducción del factor tisular en las células epiteliales colónicas. Los experimentos de cultivo de tejidos llevados aquí a cabo proporcionan evidencia de que la inducción de F3 en células de colon es un efecto directo de la toxina Shiga, pero no se puede descartar la posibilidad de que el daño a las células endoteliales por la toxina influya en la expresión de este gen in vivo.
En conjunto, todos estos hallazgos revelan que la toxina Shiga de Eschericha coli enterohemorrágica da forma poderosa a la respuesta del huésped a la infección. Aunque la toxina no contribuye a la capacidad de la bacteria para colonizar el intestino de conejos lactantes, sí lo hace en otros modelos animales y es posible que su modulación en la expresión génica del huésped mejore la capacidad del patógeno para colonizar el intestino humano. También es posible que la inflamación impulsada por Shiga aumente la diarrea y, por lo tanto, mejore la diseminación de patógenos. De manera similar, la configuración de la respuesta inmunitaria innata temprana a la infección por la toxina, probablemente tenga implicaciones para el desarrollo de respuestas inmunitarias adaptativas y la capacidad del huésped para eliminar la infección. Además, los patrones de expresión génica alterados durante la infección relacionados con la coagulación y la inflamación, sugieren una «tromboinflamación«, un mecanismo en el que la trombosis y la inflamación se unen para contribuir a la patología de la enfermedad.
Finalmente, se supone que la activación de las células inmunitarias en el colon por la toxina Shiga es fundamental para iniciar la patología en los riñones, y se han sugerido terapias anti-citocinas y antiinflamatorias como una forma de minimizar la activación de las células inmunitarias que pueden provocar daño renal. Los estudios futuros de la activación inmune provocada por el daño de la toxina Shiga en el colon podrían desbloquear nuevos objetivos para tales terapias.
Fuente de este artículo
Este artículo fue publicado originalmente en la revista “PLOS Pathogens”, bajo la referencia: Warr AR, Kuehl CJ, Waldor MK (2021) Shiga toxin remodels the intestinal epithelial transcriptional response to Enterohemorrhagic Escherichia coli. PLoS Pathog 17(2): e1009290. https://doi.org/10.1371/journal.ppat.1009290 ; y el cual está disponible con acceso libre en la propia web de PLOS Pathogens.


