
Un sistema de inteligencia artificial (IA) desarrollado por la empresa DeepMind, propiedad de Google, ha resuelto buena parte de uno de los problemas más importantes de la biología. AlphaFold logró predecir el plegado de las proteínas, lo cual está considerado como el segundo código secreto de la vida y tras el cual los científicos llevan detrás 50 años.
El primer código de la vida, es el genoma, el cual contiene toda la información para formar cualquier ser vivo. En sus letras, están todas las instrucciones para fabricar proteínas, que son las verdaderas obreras de la vida, encargadas de que alguien pueda respirar, moverse, pensar, vivir. La estructura básica del código genético es bidimensional, pero las proteínas que forma, son tridimensionales, como la diferencia entre el plano de una casa y la casa de verdad.
Además, cada proteína tiene una estructura tridimensional única que está íntimamente relacionada con su función. Sin esa estructura, sin esa forma característica, las proteínas no son nada. Asimismo, el más pequeño reordenamiento de las cadenas de aminoácidos que forman estas moléculas, puede tener efectos devastadores en nuestra salud. Las proteínas mal construidas, mal plegadas sobre sí mismas, son la causa de enfermedades tan terribles como el alzhéimer, el parkinson y muchas otras. Por tanto, conocer la estructura de las proteínas es muy importante. Si se sabe la forma de las proteínas, es más fácil entender su función y también cómo bloquearlas con fármacos, en caso necesario.
Las proteínas son moléculas grandes y complejas y cuanto más grandes, más difíciles son de modelar, ya que hay más interacciones entre los aminoácidos a tener en cuenta. En teoría, cada secuencia de aminoácidos puede dar lugar a un número innombrablemente alto de estructuras, pero en la práctica, sin embargo, cada secuencia se pliega de una única manera y lo hace muy rápido. Tal y como lo demuestra la paradoja de Levinthal, se necesitaría más que la edad del universo conocido para enumerar al azar todas las configuraciones posibles de una proteína típica antes de alcanzar la verdadera estructura 3D; sin embargo, las proteínas mismas se pliegan espontáneamente, en milisegundos.
Predecir cómo estas cadenas se plegarán en la intrincada estructura 3D de una proteína, es lo que se conoce como el «problema del plegamiento de proteínas», un desafío en el que los científicos han trabajado durante décadas. Este problema no resuelto, ya ha inspirado innumerables desarrollos, desde estimular los esfuerzos de IBM en supercomputación (BlueGene), nuevos esfuerzos de ciencia ciudadana, hasta nuevos dominios de ingeniería, como el diseño racional de proteínas.
Durante las últimas cinco décadas, los investigadores han podido determinar formas de proteínas en laboratorios utilizando técnicas experimentales como microscopía crioelectrónica, resonancia magnética nuclear y cristalografía de rayos X, pero cada método depende de una gran cantidad de prueba y error, que puede tomar años de trabajo y cuestan decenas o cientos de miles de dólares por estructura proteica. En cambio, hoy es relativamente sencillo hallar la secuencia lineal de las piezas químicas que componen la proteína, los aminoácidos, y por eso, el sueño de los biólogos es aprender a traducir la secuencia lineal en estructura tridimensional.
Es por eso, que están recurriendo a los métodos de Inteligencia Artificial (IA) como una alternativa a este largo y laborioso proceso para proteínas difíciles. La capacidad de predecir computacionalmente la forma de una proteína a partir de su código genético solo en lugar de determinarla a través de costosa experimentación, podría ayudar a acelerar la investigación.
En los años 80 y 90, los ordenadores entraron a escena, logrando avances modestos. Para estimular el área, en 1994, el biólogo computacional John Moult, de la Universidad de Maryland (EE.UU.), creó CASP (siglas en inglés de Evaluación Crítica de Predicción de Estructuras), un concurso bienal en el que grupos de todo el mundo ponen a prueba sus programas de predicción de estructuras. El reto, consiste en que, en cada edición, cristalógrafos anuncian, pero no publican, las estructuras que van resolviendo, y mientras tanto, los bioinformáticos ponen a trabajar sus programas para tratar de predecirlas. Ganan, obviamente, las predicciones más próximas al resultado experimental.
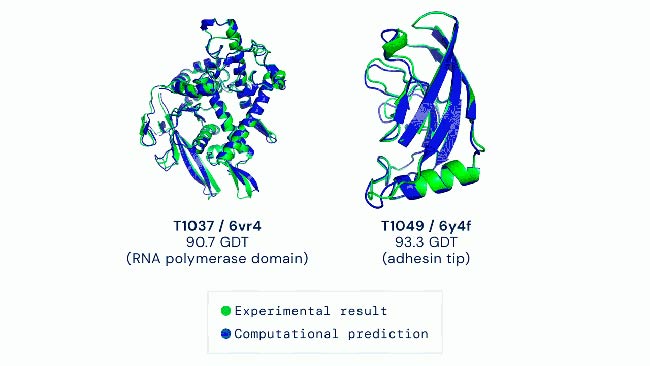
El trabajo de DeepMind en este problema, resultó en AlphaFold, que enviaron a CASP13 (2018) por primera vez y, gracias al cual, lograron un «progreso sin precedentes en la capacidad de los métodos computacionales para predecir la estructura de las proteínas», situándolos en primer lugar en la clasificación entre los equipos que ingresaron.
Y ahora, en el último concurso CASP, DeepMind compitió con otros 100 equipos en ser el mejor en predecir la forma de 100 proteínas basándose en su secuencia de aminoácidos, en donde la inteligencia artificial de Google arrasó. Su sistema, AlphaFold logró predecir el plegado de las proteínas. En concreto, AlphaFold 2 predijo correctamente la estructura de dos tercios de todas las proteínas, con una resolución equivalente a la de los métodos convencionales basados en cristalografía y microscopía, que requieren máquinas de unos ocho millones de euros y en ocasiones años o décadas de trabajo. Y, según los organizadores, la precisión de las predicciones estructurales del tercio restante es también muy alta (aunque no llega a ese nivel).
AlphaFold 2 pudo hacer en cuestión de días, lo que podría llevar años en un laboratorio, y únicamente alimentándose de información sobre las estructuras de las proteínas que ya se sabían. Es un resultado nunca alcanzado antes por ningún equipo humano ni por ninguna otra inteligencia artificial. El margen de error de las predicciones de Google es equivalente al diámetro de un solo átomo.
“Su puntuación es muy, muy superior a la de cualquier otro equipo del certamen”, explica Tim Hubbard, bioquímico del King’s College de Londres y organizador de este concurso académico durante 10 años. “Esta contribución es fundamental porque nos ayuda a resolver un problema que determina no solo la biología humana, sino la del resto de seres vivos del planeta”, resalta Hubbard.
Estos emocionantes resultados, abren la posibilidad de usar la predicción computacional de estructuras como una herramienta clave en investigación científica. Su método puede ser especialmente útil para algunas clases importantes de proteínas, como las de membrana, que son muy difíciles de cristalizar y cuya estructura es, por tanto, compleja de determinar experimentalmente.
Asimismo, el nuevo programa ayudará a desvelar la función de miles de proteínas humanas, cuya estructura aún se desconoce, así como a entender por qué bastan pequeñas variaciones en algunas proteínas para causar enfermedades. Además, señalan que “los beneficios potenciales no se limitan solo a la salud: comprender el plegamiento de proteínas ayudará en el diseño de proteínas, lo que podría desbloquear una gran cantidad de beneficios. Por ejemplo, los avances en las enzimas biodegradables, que pueden ser habilitadas por el diseño de proteínas, podrían ayudar a manejar contaminantes como el plástico y el aceite, ayudándonos a descomponer los desechos de maneras que sean más amigables con nuestra en medio ambiente”.

AlphaFold, viene siendo la culminación de varios años de trabajo y se basa en décadas de investigación previa, utilizando grandes conjuntos de datos genómicos para predecir la estructura de las proteínas.
AlphaFold 2, además del aprendizaje automático, incorpora información adicional sobre la física y la geometría del proceso de plegamiento. Su abordaje tiene aspectos novedosos, como que, en lugar de predecir las relaciones entre aminoácidos, las redes neuronales predicen directamente la estructura final jugando con imágenes. Es un sistema más complejo que “produce estructuras de proteínas mucho más precisas”, indican los autores en el libro de resúmenes de CASP. “Nuestro equipo se centró específicamente en el problema de modelar formas objetivo desde cero, sin utilizar proteínas previamente resueltas como plantillas. Logramos un alto grado de precisión al predecir las propiedades físicas de una estructura de proteína y luego usamos dos métodos distintos para construir predicciones de estructuras de proteína completas, basados en redes neuronales profundas que están capacitadas para predecir las propiedades de la proteína a partir de su secuencia genética”, aclaran desde DeepMind.
AlphaFold se basa en algoritmos que aprenden por sí mismos y ven patrones no visibles para humanos, pues están escondidos en inabarcables bases de datos con miles de proteínas compuestas por millones de fragmentos. Por eso, aunque este sistema pudiese hablar y nos contara cómo lo ha hecho, no entenderíamos la física que hay detrás de una proteína plegándose. Solo conocemos el resultado. Google no proporciona el software y esta es la parte frustrante del logro porque no va a beneficiar directamente a la ciencia.
David de Sancho, experto en estructura de proteínas en la Universidad del País Vasco, opina que el programa ha resuelto “la predicción de estructuras”, “no el plegamiento de proteínas tal como lo entiende la mayoría de quienes trabajan en el área. Esto último implicaría predecir los pasos que sigue la proteína para pasar del estado desplegado al plegado”, algo que nadie ha conseguido».
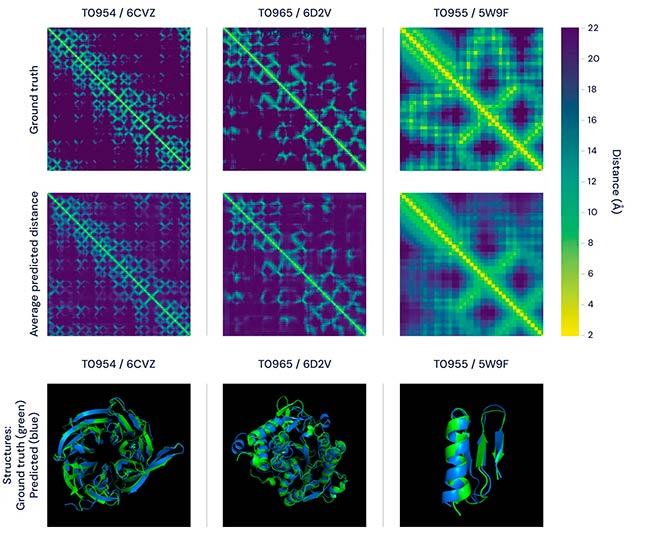
“Es un paso muy importante, se llevaba muchos años intentando descifrar ese código oculto de la vida”, resume Fernando Corrales, investigador del Centro Nacional de Biotecnología del CSIC. “En la naturaleza, puede haber decenas de miles de proteínas diferentes y cada una es un mundo completamente diferente del otro. Hay ocasiones en las que se puede conocer la estructura completa en apenas un mes, y otras en las que es complejísimo y puede llevar años o décadas”, señala Corrales. Para este científico, la inteligencia artificial de Google no dejará sin trabajo a los cristalógrafos y expertos en microscopía a corto plazo, pues aún son necesarias para corroborar las predicciones de la inteligencia artificial. Además, el problema del plegamiento de proteínas no se ha resuelto del todo, pues quedan niveles adicionales de complejidad. Las proteínas suelen funcionar en grupos formando complejos y entender su forma y movimiento es un problema que por ahora sigue sin resolver.
“Aunque de alguna manera hemos resuelto el problema de plegado de las proteínas, hay muchos otros que nos gustaría afrontar. Somos optimistas en que este sistema puede ayudarnos a comprender enfermedades y a descubrir nuevos fármacos”, señala DeepMind
Fuentes: DeepMind, El País y Xataka


