Los mosquitos transmiten muchos virus, como los que causan el dengue, introduciéndolos en nuestro organismo a través de la saliva que depositan en nuestra piel al picarnos. Dado que el dengue ha evolucionado para aprovechar la biología del mosquito y potenciar su transmisión a los humanos, comprender su ciclo de transmisión es fundamental para controlar la propagación de esta enfermedad. Los autores de este artículo, informan del descubrimiento de altos niveles de un tipo de ARN antiinmune del dengue en la saliva de los mosquitos infectados, el cual es liberado en las células del sitio de la picadura para inhibir la inmunidad innata de la persona y así potenciar la transmisión de dicho virus.
Presentación
“The anti-immune dengue subgenomic flaviviral RNA is present in vesicles in mosquito saliva and is associated with increased infectivity” (“El ARN flaviviral subgenómico antiinmune del dengue está presente en vesículas en la saliva del mosquito y está asociado con una mayor infectividad”), es un artículo científico elaborado por Yeh, S-C., Strilets, T., Castillo, D., Medkour, H., Rey-Cadilhac, F., Serrato-Pomar, I.M., Rachenne, F., Chowdhury, A., Chuo, V., Azar, S., Kiran Singh, M., Hamel, R., Missé. D., Manjunatha Kini, R., Kenney, L.J., Vasilakis, N., Marti-Renom, M. A., Nir, G., Ponpon, J., y Garcia-Blanco, M.A.; y publicado el 30 de Marzo de 2023 en el Volumen 19 de la revista PLoS Pathogens, en el cual sus autores informan del descubrimiento de un tipo de ARN antiinmune del dengue en la saliva de los mosquitos infectados por el virus, el cual sería liberado en las células del lugar de la picadura para inhibir la inmunidad innata humana y así potenciar la transmisión de dicho virus.
Los mosquitos transmiten muchos virus, como los que causan el dengue, introduciéndolos en nuestro organismo a través de la saliva que depositan en nuestra piel al picarnos. De hecho, la mitad de la población mundial corre el riesgo de contraer el virus del dengue (DENV), transmitido principalmente por el mosquito Aedes aegypti (L., 1762); por lo que existe gran interés en identificar factores que favorecen la transmisión en la saliva del mosquito para contrarrestarlos.
La transmisión del virus del dengue por mosquitos a humanos comienza con la infección de las células cutáneas del sitio de la picadura. Los científicos que estudian el comportamiento de picadura y las propiedades de la saliva de los mosquitos, han concluido que la saliva puede contener componentes que promueven la infección viral. En este estudio, los autores demuestran que cuando el virus del dengue infecta a mosquitos, se pueden encontrar en su saliva no solo los virus del dengue esperados, sino también un producto del virus que puede debilitar nuestros sistemas de defensa antiviral. Este producto se trata de un tipo de ARN antiinmune del dengue: el ARN no codificante flaviviral subgenómico antiinmune (ARNsf), el cual, localizándose en las vesículas extracelulares salivares del mosquito, se liberaría en las células del sitio de la picadura, para inhibir la inmunidad innata humana y potenciar la transmisión del virus del dengue.
En concreto, este ARN antiinmune del dengue, es producido a partir de la degradación parcial del genoma del ARN flaviviral (ARNg) por las exorribonucleasas 5’-3’ del hospedador, que se bloquean en estructuras secundarias de la región no traducida (UTR) 3’; y es capaz de inhibir la respuesta inmunitaria tanto en células humanas como de insectos, por lo que se suele referir a él como un mediador antiinmunitario. Cabe indicar que también existe este tipo de ARN en otros virus similares como el Zika.
Materiales y Métodos
A continuación, se resumen los métodos de trabajo llevados a cabo por los científicos para demostrar su hipótesis de que existe ARN antiinmune del dengue en la saliva de los mosquitos que se libera en la picadura e inhibe la respuesta innata, favoreciendo la propagación del virus.
Líneas celulares, virus y mosquitos
Las líneas celulares C6/36 (CRL-1660) de Aedes albopictus (Skuse 1895) y riñón de cría de hámster BHK-21 (CCL-10), se cultivaron en medio RPMI, suplementado con 10% de suero fetal bovino (SFB) inactivado por calor y 1% de penicilina-estreptomicina a 28°C y 37°C, respectivamente, en 5% de CO₂.
La línea celular Huh-7 (clon JTC-39) se obtuvo de la Fundación Japonesa de Ciencias de la Salud, Osaka, y se mantuvo en medio Eagle modificado de Dulbecco (DMEM), suplementado con 10% de SFB, 1% de aminoácidos no esenciales y 1% de penicilina-estreptomicina a 37°C en 5% de CO₂.
Los fibroblastos dérmicos humanos neonatales (NHDF) (CC-2509, Lonza), se cultivaron a 37°C con 5% de CO₂ en medio basal de crecimiento de fibroblastos (FBM, Lonza) suplementado con el kit de medio de crecimiento de fibroblastos 2 (FGM-2, Lonza) y 2% de SFB.
La cepa C de Nueva Guinea (NGC) del virus del dengue 2 (DENV2) se obtuvo de la ATCC (VR-1584) y del Centro Mundial de Referencia para Virus y Arbovirus Emergentes (WRCREVA) de la UTMB; mientras que las cepas de bajo pase de las cepas PR1940, PR6452 y PR9963 del DENV2, fueron donadas por la División de Dengue de los Centros para el Control y la Prevención de Enfermedades (CDC) de San Juan, Puerto Rico, y se obtuvieron del laboratorio del Prof. Eng Eong Ooi en la Facultad de Medicina de Duke-NUS.
Las cepas del DENV2 se propagaron en las C6/36, se titularon mediante ensayo de placa con células BHK-21 y se almacenaron a -80°C.
Por otro lado, la colonia de Aedes aegypti (SGP) de Singapur se estableció y crió en el insectario Duke-NUS. La colonia de Ae. aegypti Rockefeller (ROCK) fue criada utilizando métodos estándar por el Centro de Servicios de Insectarios de la UTMB. Y la colonia de Ae. aegypti (BORA) de Bora-Bora se crió en el insectario VectoPole en MIVEGEC.

Infección oral de mosquitos
Mosquitos hembra SGP de tres a cinco días de edad se mantuvieron en ayunas durante 17h y se les ofreció una dieta con sangre que contenía 40% de eritrocitos lavados de sangre de cerdo libre de patógenos específicos, 5% de ATP 10mM, 5% de suero humano, 50% de medio RPMI y 107 unidades formadoras de placa (ufp) por ml en todos los experimentos.
Para la infección se utilizaron células NGC de las cepas ATCC, PR1940, PR6452 y PR9963. Los mosquitos se expusieron a la dieta con sangre durante 1,5h y se seleccionaron hembras completamente ingurgitadas, que se mantuvieron con agua y una solución de azúcar al 10% hasta su análisis a los 10 o 15 días después de la dieta con NGC o PR, respectivamente.
Inoculación de mosquitos
Mosquitos hembra ROCK de tres a cinco días de edad se anestesiaron con frío y se inocularon con 180ufp de DENV2 NGC de WRCEVA (UTMB). Mosquitos hembra BORA de tres a cinco días de edad se anestesiaron con frío y se inocularon con 50ufp de DENV2 NGC de ATCC. Los mosquitos control se inocularon con el mismo volumen de PBS.
Tras la inoculación, los mosquitos se mantuvieron durante 10 o 7 días con acceso ad libitum a una solución de sacarosa al 10%.
Recolección de saliva de mosquitos
Los mosquitos hambrientos se inmovilizaron cortándoles alas y patas. Las probóscides de mosquito se insertaron individualmente en puntas de 20μl que contenían 10μl de medio precalentado con 0,5mM de ATP durante 30 minutos. Para identificar a los mosquitos que ingirieron rodamina y, por lo tanto, salivaron, el medio fue RPMI suplementado con rodamina al 0,01%.

Establecieron la presencia del ARN antiinmune del dengue en la saliva de los mosquitos mediante tres métodos diferentes: Northern blot, RT-qPCR y secuenciación de ARN.
Northern blot
Para los Northern blot utilizaron el kit NorthernMax (Ambion) con modificaciones al protocolo del fabricante. Se extrajo el ARN total de 50 mosquitos infectados por vía oral mediante RNAzol RT y se separó en un gel desnaturalizante con acrilamida al 5% y urea 8M. También se cargó en el gel una escalera de ARN monocatenario biotinilado.
El ARN se transfirió a una membrana de nailon (Hybond-N; Biodyne B) mediante Trans-Blot Turbo (Bio-Rad) a 25V durante 30 min. La membrana se reticuló con UV, se prehibridó y se hibridó con una sonda de dsADN marcada con biotina-16-dUTP, que incluía el 3’UTR completo, y se generó utilizando los cebadores. Tras los lavados y el bloqueo, la membrana se tiñó con estreptavidina IRDYE 800cw. Las imágenes se tomaron con el sistema de imágenes Odyssey CLx (LI-COR).
Por otro lado, el ARN total de la saliva de 30 mosquitos inoculados con DENV2 y simulacro (PBS), se extrajo utilizando Trizol LS de acuerdo con el protocolo del fabricante con las siguientes modificaciones: el ARN se precipitó con 1μL de reactivo GlycoBlue a -20°C durante 2h. El ARN total extraído se mezcló con colorante de carga de ARN 2X y se separó en geles de TBE-Urea de acrilamida al 6% a 110V durante 2,5h con TBE 1X. También se cargó en el gel una escalera de ARN monocatenario biotinilado. Como control para visualizar el ARNsf, utilizaron ARN celular total de células Huh-7 no infectadas, junto con ARN total de células Huh-7 infectadas con DENV2 NGC, con un MOI de 2, a las 48 horas posteriores a la infección.
El gel se transfirió a una membrana de nailon Biodyne B utilizando el módulo XCell II Blot a 15V durante 1,5h en TBE 0,5X. La membrana se reticuló mediante UV y se prehibridó como se describió previamente. Para visualizar el ARNf, se utilizó 1µg de sonda de dsADN marcada con biotina 16-dUTP, generada con los cebadores. La membrana se hibridó a 78°C durante la noche. Tras la hibridación, se realizaron dos lavados de baja astringencia a temperatura ambiente durante 10 min cada uno. Todos los demás lavados se realizaron según el protocolo NorthernMax (Ambion). Finalmente, la membrana se tiñó con estreptavidina IRDYE 800cw y se obtuvieron imágenes con el sistema de imágenes Odyssey CLx (LI-COR).
Cuantificación absoluta de copias de ARNg y ARNsf1 mediante RT-qPCR en tiempo real
Para este caso, utilizaron mosquitos infectados por vía oral. Sus glándulas salivales se homogeneizaron con microesferas de sílice de 1mm mediante el mini-beadbeater-96 en 350μl de tampón de lisis TRK, y las mezclas con saliva se lisaron en 350μl de tampón de lisis TRK. El ARN total se extrajo con el kit E.Z.N.A. Total RNA I (Omega Bio-Tek). El ARNg y el 3’UTR/ARNsf1 se cuantificaron en números absolutos mediante RT-qPCR de un solo paso y curvas estándar.
Para calcular el límite de detección (LdD), los fragmentos de ARNg y 3’UTR/ARNsf1 que abarcan las dianas de qPCR y generados para las curvas estándar, se diluyeron en serie 10 o 2 veces, de 1,2 x 10⁻¹ a 1,9 copias para el ARNg, y de 6 x 10⁻¹ a 117 copias para el ARNsf1. Se cuantificaron cuatro repeticiones técnicas por dilución de tres repeticiones independientes mediante RT-qPCR de un solo paso. Las fracciones de réplicas positivas por copia de ARNg o 3’UTR/ARNsf1 se representaron gráficamente frente a una curva sigmoidea para identificar el LdD con un 95% de confianza.
El número de copias de ARNsf1 se calculó restando el número de fragmentos de ARNg1 y 3’UTR (estimado mediante los cebadores 3’UTR/ARNsf1) al número de fragmentos de ARNg (estimado mediante los cebadores de envoltura). Para las muestras infectadas con una cantidad detectable de ARN antiinmine del dengue, la relación ARNsf:ARNg se calculó dividiendo la cantidad de ARNsf entre la cantidad de ARNg. La tasa de detección de ARNg correspondió al número de muestras con una cantidad detectable de este sobre el número total de muestras analizadas.
Secuenciación de ARN antiinmune del dengue
Se extrajo el ARN total de 50 mosquitos enteros o 10 muestras de saliva recolectadas tras la infección oral mediante RT con RNAzol. Los fragmentos de ARN antiinmune del dengue se circularizaron utilizando la ARN ligasa 1 T4 a 4°C durante la noche, se transcribieron de forma inversa utilizando SuperScript III con el cebador 5’-GCTGTTTTTTGTTTCGGG-3’, ubicado en los nucleótidos 10621-10604 del genoma de NGC DENV2, y se amplificaron utilizando el cebador directo 5’-AAAATGGAATGGTGCTGTTG-3’ (nucleótidos 10690-10709 del genoma de NGC) y el cebador utilizado para la transcripción inversa.
Los productos de PCR se separaron en gel y las bandas de tamaño esperado se clonaron con el kit Topo TA antes de la secuenciación (1.ª base).
Ensayo de resistencia a la ARNasa tras tratamientos con detergente o proteinasa
Se agruparon veinte muestras de saliva recolectadas de mosquitos con o sin infección oral por NGC y se dividieron en cuatro fracciones de igual volumen. Cada subconjunto se utilizó para analizar una de las cuatro combinaciones de ARNasa con tratamientos de Triton X-100 o proteinasa K (PK).
El ARNf1 transcrito in vitro, generado para la curva estándar, se añadió a cada subgrupo de saliva no infectada. Las muestras se trataron con Triton X-100 al 0,1% a temperatura ambiente durante 30 minutos antes de añadir 3μl de ARNasa A/T1 a 37°C durante 30 minutos. Para dispersar la solución de Triton X-100 manteniendo la integridad de las vesículas extracelulares, se pipeteó suavemente la mezcla de tratamiento diez veces. Las muestras se trataron con 0,25μg de PK a temperatura ambiente durante 30 min y con 2mM de fluoruro de fenilmetilsulfonilo (PMSF) a temperatura ambiente durante 10 min para inhibir la PK, también antes de añadir 3μl de ARNasa A/T1 a 37°C durante 30 min. Dado que el PMSF se disolvió en DMSO, se añadió DMSO al 1% como control cuando no se añadió PMSF. El ARN se extrajo con el minikit QIAamp viral RNA y se cuantificaron el ARNf y el ARNg.
Microscopía electrónica de transmisión (TEM) de la saliva del mosquito Ae. aegypti
10µL de saliva de mosquitos no infectados (simulados) e infectados con DENV2 se absorbieron directamente en rejillas de cobre Formvar durante 10 minutos y se eliminó el exceso de saliva. A continuación, las rejillas se fijaron flotando en una mezcla de glutaraldehído al 2% y paraformaldehído al 2% durante 10 minutos y se eliminó el exceso de fijador.
A continuación, las rejillas se flotaron en una solución de acetato de uranilo y ácido oxálico 0,15M en una proporción 1:1 durante 5 minutos y se eliminó el exceso de oxalato de uranilo. Finalmente, las rejillas se embebieron mediante flotación en una solución que contenía 2% de metilcelulosa y 4% de acetato de uranilo en una proporción de 9:1 durante 10 minutos en hielo.
Finalmente, las rejillas se examinaron en un microscopio electrónico de transmisión JEOL JEM-1400 (EE. UU.) a 80 kV. Las imágenes se adquirieron con una cámara CCD Orius SC2001 (Gatan, Pleasanton, CA) montada en la base.

Diseño de la biblioteca Oligopaints
Utilizando la metodología Oligominer, se diseñó la biblioteca de sondas Oligopaints RNA FISH para unirse al genoma de NGC DENV2. Para ello, se utilizó el script blockParse para seleccionar oligos de entre 35 y 41nt de longitud con una temperatura de fusión de 42 a 47°C. Los oligos seleccionados se alinearon con los genomas de DENV2 NGC (Homo sapiens) y Ae. Aegypti, utilizando bowtie2. Los oligos candidatos se filtraron adicionalmente utilizando el script kmerFilter del algoritmo JellyFish. Por último, los oligos que se unían a los complementos inversos del genoma de DENV2 se generaron con el script probeRC. En total, la biblioteca constó de 240 oligos con una densidad promedio de 22 oligos/kb del genoma de DENV2.
Las secuencias de la calle principal y de la calle secundaria se anexaron a los extremos 5′ y 3′ (respectivamente) de la biblioteca de Oligopaints de DENV2 mediante la herramienta de anexión OligoLego. 233 oligos que se unieron a los nucleótidos 1-10 293 del genoma NGC de DENV2 se codificaron como ARNg, mientras que 7 oligos que se unoeron a los nucleótidos 10 294-10 717 se codificaron como 3’UTR.
Recubrimiento con APTES de portaobjetos con cámara de vidrio ibidi
Portaobjetos μ-Slide de 8 pocillos con fondo de vidrio (ibidi) se lavaron con 200μL de acetona, se trataron con KOH 3M y se sonicaron durante 1 hora. Tras los lavados con agua, cada pocillo se incubó con 200μL de 3-aminopropiltioetoxisilano (APTES) al 2% (vol/vol) en acetona seca durante 30s. Los portaobjetos se lavaron dos veces con 400μL de agua y se secaron durante 3 horas a 37°C.

Tinción FISH de ARN con Oligopaints
Se sembraron 2 x 10⁻¹ células C6/36 en portaobjetos y se infectaron con NGC DENV2 de WRCEVA a una MOI de 0,3 durante 1 hora. Las células control se infectaron simuladamente con PBS.
Tras la infección, las células se mantuvieron en medio RPMI suplementado con 5% de FBS, HEPES 0,01M (pH 7,4) y 1% de penicilina/estreptomicina durante 2 o 24 horas. Tras la incubación, se retiró el medio y las células se enjuagaron dos veces con PBS 1X.
Los grupos de saliva recogidos de los mosquitos inoculados se secaron en portaobjetos recubiertos con APTES durante 20 minutos. Los grupos de células y saliva se fijaron con paraformaldehído al 4% durante 10 minutos, se permeabilizaron con Triton X-100 al 0,5% durante 15 minutos, se bloquearon con BSA al 5% durante 1 hora, se lavaron con Tween-20 al 1% y se enjuagaron con Tween-20 al 1% en tampón de citrato de sodio salino 2X (SSC).
Se añadió la solución de hibridación primaria, compuesta por Tween-20 al 1% en 2X SSC, sulfato de dextrano al 10%, formamida al 50%, 200μg/μL de ADN de esperma de salmón cortado (Stratagene), 100μg/μL de ARNt de levadura y 4μM de la biblioteca de sondas FISH Oligopaints DENV2 3’UTR/gRNA.
Las muestras se incubaron en un baño de agua a 60°C durante 3 min. Se lavaron varias veces con Tween-20 al 1% en 2X SSC precalentado (60°C), dos veces con Tween-20 al 1% en 2X SSC a TA y una vez con PBS a TA.
Se añadió la solución de hibridación secundaria, compuesta por 2X SSC, 0,03% de Tween-20, 30% de formamida, 5% de sulfato de dextrano, 200μg/μl de ADN de esperma de salmón cortado, 100μg/μl de ARNt de levadura, 0,5μM de oligonucleótidos puente, 0,5μM de oligonucleótidos secundarios conjugados con ATTO 565 y Alexa Fluor 647.
Las muestras se incubaron durante 30 min a temperatura ambiente. Se lavaron cuatro veces durante 5 min con 1% de Tween-20 en 2X SSC y 35% de formamida. Las muestras de C6/36 se colocaron en un tampón de imagen compuesto por 4mM de ácido protocatéquico (PCA), 15nM de protocatecuato 3,4-dioxigenasa (PCD) y 1,5mM de ácido 6-hidroxi-2,5,7,8-tetrametilcroman-2-carboxílico, con la adición de 300nM de 4,6-diamidino-2-fenilindol (DAPI). Las muestras de saliva de mosquito se colocaron en el mismo tampón sin DAPI.
La obtención de imágenes confocales se realizó en un sistema Leica TCS SP8 STED 3X, equipado con un láser de luz blanca pulsada sintonizable (470-670nm) y un láser de diodo UV de 405nm para excitaciones a 405nm, 564nm y 647nm. La emisión de DAPI, ATTO 565 y Alexa Fluor 647 se monitorizó en el rango de 413-450nm, 568-640nm y 653-710nm, respectivamente, utilizando el divisor de haz acústico-óptico integrado en combinación con un sistema de detección espectral multibanda sintonizable basado en prismas del sistema confocal. Las imágenes se adquirieron con un objetivo de inmersión en aceite Leica HC PL APO CS2 100×/1.4.

Transcripción y transfección in vitro de la 3’UTR PR6452 de DENV2
La 3’UTR de DENV2 se transcribió in vitro a partir de un vector pcDNA 3.1-Apt-PR6452-3’UTR que contiene la secuencia del promotor de la ARN polimerasa T7, la secuencia del aptámero de ARN de estreptomicina/tobramicina y la secuencia de la 3’UTR de DENV2 PR6452.
La 3’UTR pcDNA 3.1-Apt-PR-3’UTR se linealizó con XbaI (NEB), se transcribió con el kit de síntesis de ARN HiScribe T7 (NEB), se trató con ADNasa I y se purificó mediante extracción con fenol-cloroformo. La integridad del ARN se evaluó en un gel de agarosa desnaturalizante al 1%.
Las células C6/36 se transfectaron con 1μg de ARN 3’UTR utilizando Lipofectamina 3000. Dos horas después de la transfección, las células se enjuagaron dos veces con PBS 1X, se fijaron durante 10 minutos con paraformaldehído al 4% y se procesaron para ARN-FISH, como se describió anteriormente.
Cuantificación de las proporciones 3’UTR:ARNg a partir de imágenes
Para segmentar cada célula C6/36 en las imágenes e incluir la señal de los núcleos interno y externo en el análisis, añadieron proyecciones de máxima intensidad de las sondas 3’UTR, las sondas ARNg y las pilas de imágenes DAPI. Aplicaron un filtro máximo para rellenar las áreas vacías dentro de cada célula, seguido de un filtro gaussiano para suavizar las imágenes.
Las imágenes resultantes se convirtieron en máscaras binarias y se utilizaron para identificar el área ocupada por cada célula individual mediante ImageJ. Para garantizar que solo se analizaran las células infectadas, midieron la densidad máxima integrada de ARNg en el conjunto de células simuladas (no infectadas) y utilizaron este valor para descartar células en las imágenes infectadas.
Las proporciones 3’UTR/ARNsf:ARNg se calcularon colapsando las pilas de imágenes a proyecciones de máxima intensidad y dividiendo las densidades integradas en cada célula segmentada (suma de los valores de los píxeles de la selección). Esto se realizó con la macro ImageJ “segment_and_measure_cells.ijm”.
En el caso de las imágenes 3’UTR/ARNsf con señal de autofluorescencia nuclear, utilizaron las densidades integradas citoplasmáticas de las señales de ARNg y 3’UTR/sARNsf para el análisis. Mediante ImageJ, segmentaron manualmente el área nuclear con las imágenes DAPI y vaciaron el área ocupada por el núcleo en las pilas de imágenes 3’UTR/ARNsf y ARNg. Una vez eliminadas, las proporciones 3’UTR/ARNsf:ARNg se calcularon como se describió anteriormente.
Para segmentar cada vesícula extracelular, identificaron el mejor corte z enfocado en pilas de imágenes de campo amplio y segmentaron manualmente el área de la vesícula con ImageJ. Esto se realizó con la macro de ImageJ “segment_vesicles_best_focus.ijm”. La relación 3’UTR/ARNsf: ARNg se calculó igual que para las células utilizando la macro ImageJ “measure_vesicles_best_focus.ijm”.

RT-PCR de DENV2 de saliva de mosquito
El ARN total de la saliva de mosquito y de los sobrenadantes C6/36 infectados se extrajo con Trizol LS. El ADNc se sintetizó con el kit de transcripción inversa de ADNc de alta capacidad, y la qPCR se realizó con la mezcla maestra para PCR SYBR Green y el instrumento StepOne Plus, utilizando cebadores.
Las muestras se cargaron en un gel de agarosa al 1% y se tiñeron con bromuro de etidio para visualizar los productos de PCR.
Obtención de imágenes de nanoesferas Tetraspek
Sonicaron nanoesferas fluorescentes Tetraspek de 100nm durante 10 min, las diluyeron 1:5000 en PBS 1X y agitaron en vórtex. Se adhirieron 200μl de nanoesferas diluidas a portaobjetos μ-Slide de 8 pocillos con fondo de vidrio durante 1 hora.
Tras la adhesión, los portaobjetos se lavaron dos veces con PBS antes de la adquisición de imágenes.
Los diámetros aparentes de las nanoesferas se determinaron con ImageJ.
Infección de células humanas mediante incubación con saliva de mosquito infectada
Se sembraron 3000 células Huh-7 en cada pocillo de un portaobjetos con cámara extraíble de 12 pocillos (ibidi GmbH) y se cultivaron en 200μl de medio completo. Se retiró el medio y las células se incubaron con grupos de 40 salivas de mosquito infectadas por vía oral durante dos horas a 37°C. Se retiró el inóculo de saliva y se reemplazó con 200μl de DMEM suplementado con 2% de FBS.
Dos días después de la infección, las células se fijaron con paraformaldehído al 4% durante 20 min, se permeabilizaron con SDS al 0,1% en PBS durante 10 min, se bloquearon con BSA al 0,1% en PBS durante 40 min y se incubaron con anticuerpo anti-dsRNA J2 1:500 en solución de bloqueo durante 1 hora a temperatura ambiente. Después de dos lavados con PBS, las células se incubaron con anticuerpo anti-IgG de ratón marcado con Alexa Fluor 488 1:1000 en medio de bloqueo durante 1 hora. Después de los lavados, los portaobjetos se montaron con Prolong gold Antifade Mountant.
Las imágenes se tomaron con un nanoscopio TCS SP8 STED 3X (Leica). Los focos de dsRNA se detectaron y cuantificaron con ImageJ, utilizando una macro propia que procesó el fondo a 50 píxeles, ajustó el umbral para eliminar la señal de ruido y cuantificó el tamaño de partícula superior a 0,08μm².
Para comprobar si el anticuerpo anti-dsRNA J2 detectaba el ARN antiinmune den dengue, se produjo in vitro ARNsf1 de longitud completa de DENV2 NGC, como se detalló anteriormente. Se simularon 5 x 10⁻¹ células Huh-7, infectadas con DENV2 NGC de ATCC a una MOI de 1 durante 1 hora, o transfectadas con ARNsf plegado mediante TransIT-mRNA.
Las células se tiñeron 24 horas después de la infección o 1 hora después de la transfección con ARNSf, y se observaron con un microscopio confocal LSM780 (Zeiss). Para cuantificar el ARN antiinmune del dengue intracelular, se lavaron las células con PBS, se recogieron en tampón de lisis TRK, se extrajo el ARN con el kit E.Z.N.A. Total RNA I y se cuantificó el ARNsf mediante RT-qPCR, como se detalló anteriormente.
Cuantificación de la infección dérmica de fibroblastos con saliva
Se filtraron grupos de 40 muestras de saliva de mosquitos inoculados a través de filtros de 0,22μm y se almacenaron a -80°C hasta su uso.
Se cultivaron 3000 células NHDF con 200μl de medio completo para fibroblastos suplementado con 2% de SFB en un portaobjetos extraíble de 12 pocillos. Veinticuatro horas después de la siembra, se retiró el medio y las células se incubaron con 25μl de saliva diluida con 25μl de medio para fibroblastos sin SFB durante 1 hora, con agitación suave.
Las copias de ARNg de los diferentes grupos de saliva infectada se normalizaron y se utilizó saliva no infectada para ajustar el volumen total a 25μl de saliva. Se retiró el inóculo y se reemplazó con 200μl de medio para fibroblastos con 0,5% de SFB. 72 horas después de la infección, las células se recogieron en 250μl de tampón de lisis TRK y se extrajo el ARN total siguiendo las instrucciones del fabricante.
El ARNg se cuantificó mediante RT-qPCR de un solo paso utilizando el kit iTaq Universal SYBR green de un solo paso (Bio-Rad). La mezcla de reacción contenía 300nM de cebadores directos e inversos y 2μl de extracto de ARN en un volumen total de 10μl. Los ensayos de RT-qPCR se realizaron en el sistema de PCR en tiempo real AriaMx, seguidos de un análisis de la curva de fusión. Se generó una curva estándar absoluta para el ARNg y se calculó un límite de detección del 95 %.
La diana de qPCR del ARNg generada para las curvas estándar se diluyó en serie 10 o 2 veces desde 2202 hasta 0,04 copias. Se cuantificaron seis repeticiones técnicas por dilución mediante RT-qPCR de un solo paso.
Transcripción y transfección in vitro de la UTR 3’ de DENV2 NGC
La UTR 3’ de DENV2, tanto sentido como antisentido, se transcribió in vitro a partir de un producto de PCR amplificado a partir del vector pcDNA 3.1-DENV2 NGC 3’UTR que contiene la secuencia de la UTR 3’ de DENV2.
Se utilizaron 100ng de pcDNA 3.1-DENV2 NGC 3’UTR como molde para la PCR, usando los cebadores sentido y antisentido de la UTR 3’ de DENV2. La PCR se realizó utilizando la mezcla maestra Taq 2X de acuerdo con el protocolo del fabricante.
Los productos de PCR de los amplicones 3’UTR sentido y antisentido se purificaron utilizando Nucleospin Gel y PCR Cleanup Mini Kit. Los productos de PCR purificados se transcribieron in vitro con el kit de síntesis de ARN HiScribe T7 (NEB), se trataron con ADNasa I y se purificaron con extracción con fenol-cloroformo.
La integridad y especificidad del ARN se evaluaron en un gel de agarosa desnaturalizante al 1% y un tratamiento con ARNasa T1. Finalmente, los ARN 3’UTR sentido y antisentido se trataron con ARN 5′ polifosfatasa y se purificaron con el kit RNA Clean and Concentrator según el protocolo del fabricante.
Por otra parte, cultivaron 1 x 10⁻¹ células Huh-7 en una placa de 48 pocillos y los transfectaron con 50ng de ARN 3’UTR sentido y antisentido utilizando Lipofectamina 3000, y se reconstituyó con DMEM suplementado con 5% de SFB. Dos horas después de la transfección, se eliminó el medio restante y las células se infectaron con DENV2 NGC a una MOI de 0,1 en DMEM suplementado con 5% de SFB, durante 2 horas a 37°C. Después de la infección, se eliminó cualquier inóculo de virus restante y las células se mantuvieron en 200μL de DMEM suplementado con 10% de FBS, 1% de aminoácidos no esenciales y 1% de penicilina-estreptomicina.
Cuantificación absoluta de copias de ARNg de DENV2 y cuantificación relativa de transcritos de ARNm de células Huh-7 transfectadas mediante 3’UTR
17 horas después de la infección, se recogieron los sobrenadantes celulares de las células transfectadas y se centrifugaron durante 20 minutos a 3000 x g para sedimentar los restos celulares. Inmediatamente, se procesaron 140μL del sobrenadante clarificado para la extracción de ARN viral utilizando el minikit QIAamp viral RNA, según el protocolo del fabricante.
Las células transfectadas restantes se lavaron dos veces con PBS y se extrajo el ARN total utilizando 140μL de Trizol y el kit de purificación de ARN Direct-zol, según el protocolo del fabricante.
El ARN intracelular y extracelular se utilizó para la síntesis de ADNc utilizando el kit de transcripción inversa de ADNc de alta capacidad. El ADNc intracelular se diluyó 1:5, mientras que el ADNc extracelular se diluyó 1:25 con agua. Se utilizaron 2,5μL de ADNc diluido para la qPCR utilizando el iTaq Universal SYBR Green Supermix (Bio-Rad) en una reacción de 10μL. La mezcla de reacción contenía 400nM de cebadores directos e inversos para el ARNg de DENV2. La qPCR se realizó en el BioRad CFX384 Real Time System.
El número absoluto de copias del ARNg de DENV2 se determinó mediante una curva estándar, como se describió previamente.
Los niveles de transcripción de ARNm se cuantificaron mediante qPCR relativa a partir de muestras de ADNc intracelular descritas anteriormente. Se utilizaron 2,5μL de ADNc diluido para la qPCR mediante el iTaq Universal SYBR Green Supermix (Bio-Rad) en una reacción de 10μL. La mezcla de reacción contenía 300nM de cebadores directos e inversos para los IFN e ISG. La qPCR se realizó en el BioRad CFX384 Real Time System.
Análisis estadístico
Las diferencias en la relación ARNsf:ARNg, las copias de ARNg y ARNsf transformadas logarítmicamente, se analizaron mediante pruebas t de Student para muestras no pareadas. Se utilizaron comparaciones múltiples con la prueba LSD de Fisher para los ensayos de resistencia. Los focos de ARNdc y el porcentaje de células positivas para ARNdc se analizaron mediante la prueba U de Mann-Whitney. Se utilizó Prism v5 (GraphPad) para el análisis.
Para las imágenes RNA-FISH, las diferencias en la relación 3’UTR/ARNsf:ARNg se probaron utilizando una prueba U de Mann-Whitney bilateral con R. Las diferencias en los diámetros aparentes de EV se probaron utilizando pruebas t de Student bilaterales con correcciones de Welch para la prueba de varianza desigual con R. Las diferencias en los números absolutos de copias de gRNA se probaron utilizando un LSD de Fisher sin corregir utilizando Prism v9 (Graphpad). Las diferencias en los niveles relativos de ARNm de transcripciones de IFN e ISG se probaron utilizando una prueba t no pareada con corrección de Welch y la prueba de Dunn sin corregir utilizando Prism v9 (GraphPad).
Resultados del estudio sobre la presencia de ARN antiinmune del dengue en la saliva de mosquitos infectados
Y a continuación, se comentan los resultados obtenidos por los científicos tras llevar a cabo todo el trabajo descrito para estudiar la presencia de ARN antiinmune del dengue en la saliva de mosquitos infectados.
La saliva de mosquitos infectados con DENV2 contiene altos niveles de ARN antiinmune del dengue
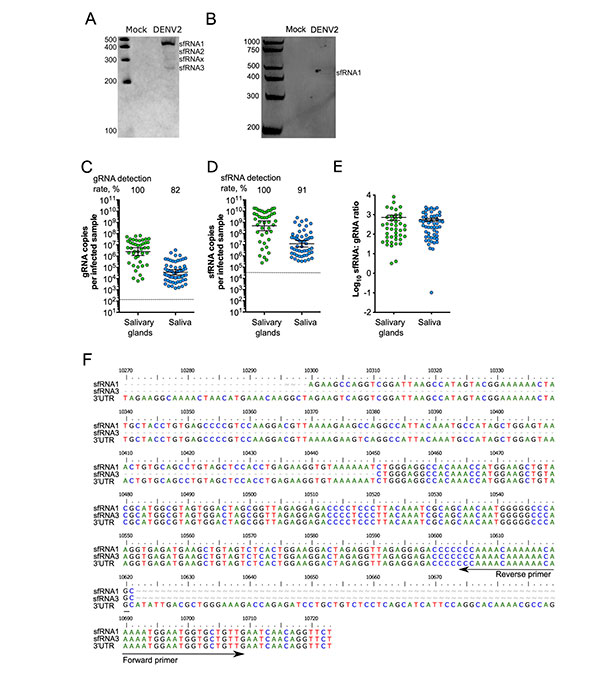
Existen varios tipos de ARN antiinmune del dengue producidos por inhibición diferencial de exonucleasas en células de mosquitos. En el ARN extraído de mosquitos Ae. aegypti enteros infectados oralmente con la cepa DENV2 NGC, detectaron tres de las especies esperadas, sfRNA1-3, y una especie adicional, sfRNAx, tal y como se ve en la Figura 1A inferior.
Para comprobar si el ARN antiinmune del dengue se secreta en la saliva de mosquitos, analizaron el ARN extraído de saliva agrupada de Ae. aegypti inoculada con DENV2 NGC mediante transferencia Northern y detectaron sfRNA1 (Figura 1B).
Dado que sfRNA1 fue la principal especie detectada en mosquitos enteros y saliva, cuantificaron las copias de sfRNA1 mediante RT-qPCR. Para tener en cuenta la variación en la eficiencia de RT y qPCR al medir ARNsf y ARNg, calcularon el número absoluto de copias de ARNsf y ARNg utilizando curvas estándar generadas a partir de ARNsf y ARNg transcritos in vitro. Además, calcularon por separado las tasas de detección y las cantidades en muestras positivas para diferenciar entre dos fenómenos. Interpretaron las tasas de ARNg como una medida del inicio de la infección, y las tasas de ARNsf como una indicación del inicio exitoso de la producción de ARN antiinmune del dengue. Y también interpretaron las cantidades de ARNg como un indicador de la capacidad del virus para replicarse, y las cantidades de ARNsf como una información sobre el proceso de producción y la estabilidad del ARN antiinmune del dengue.
A los 10 días después de la infección oral (d.p.i.), el ARNg estaba presente en las glándulas salivales y en la saliva (media geométrica de 3,6 x 104 copias por saliva infectada expectorada por un mosquito, Fig. 1C). Detectaron sfRNA1 en el 100% de las glándulas salivales infectadas y, de manera importante, en el 91% de las muestras de saliva infectadas (media geométrica de 1,1 x 107 copias de sfRNA por muestra de saliva infectada, Fig. 1D).
Para normalizar el ARNsf con su precursor y nivel de infección, calcularon las razones ARNsf:ARNg. La razón en las glándulas salivales fue de 720 (± 221,5) y no fue significativamente diferente de la razón en la saliva, que fue de 559,2 (± 89,07) (Fig. 1E).
También detectaron ARNsf en la saliva de mosquitos infectados oralmente con otras tres cepas de DENV2: PR1940, PR6452 y PR9963, lo que respalda la generalidad del ARNsf salival. Cabe destacar que las proporciones de ARNsf y ARNg varían entre cepas del virus (de 26 a 559), lo que podría estar relacionado con variaciones genéticas entre cepas o con la biología variable del mosquito.
Para validar la presencia de ARNsf en saliva infectada con un enfoque independiente, utilizaron el método consolidado de circularización y secuenciación de ARN para determinar la secuencia de las especies de ARNs. Detectaron secuencias de sfRNA1 y sfRNA3 en mosquitos infectados con NGC DENV2 y en saliva. Estos resultados les llevaron a concluir que el ARNsf, un potente inhibidor inmunitario, está presente en la saliva de mosquitos infectados con DENV2.
El ARN antiinmune del dengue está presente en compartimentos sensibles a detergentes en la saliva de mosquitos
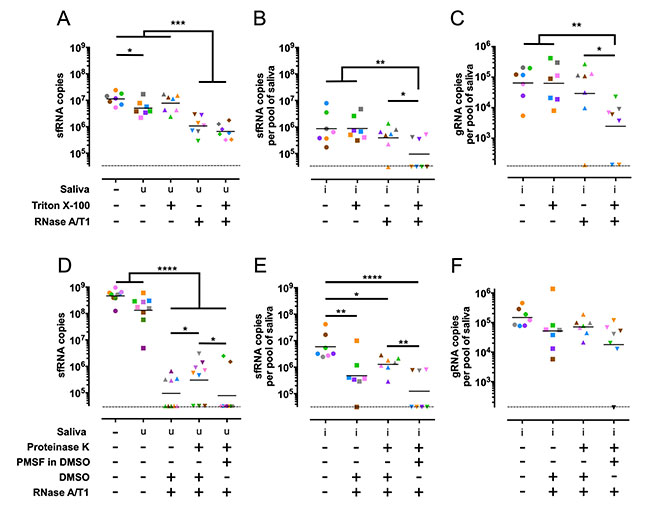
Los ARNnc extracelulares pueden empaquetarse en vesículas extracelulares (VE), incluyendo microvesículas y exosomas, o en complejos de ribonucleoproteína con proteínas de unión al ARN. Por lo tanto, para determinar cómo se secreta y protege el ARN antiinmune del dengue en la saliva, sometieron la saliva infectada a diferentes tratamientos bioquímicos y evaluaron su efecto sobre los niveles de ARNsf.
Utilizaron ARNsf1 sintetizado in vitro, que añadieron a la saliva de mosquitos no infectados, como control para estos experimentos. El ARNsf1 incubado en saliva no infectada fue relativamente estable, pero, como era de esperar, este ARN pudo digerirse añadiendo ARNasas A/T1 (Fig. 2A). Por el contrario, el ARNsf1 encontrado en la saliva infectada fue resistente al tratamiento con ARNasas (Fig. 2B). Sin embargo, si la saliva infectada se pretrató con Triton X-100, la gran mayoría del ARNsf en la saliva acabó siendo digerido por la ARNasa (Fig. 2B, en donde cada color representa una repetición independiente en el tratamiento con ARNasa).
El ARNg en la saliva también fue resistente a la ARNasa, a menos que se pretratara con Triton X-100 (Fig. 2C), presumiblemente porque está empaquetado en viriones y VE. Los ensayos indicaron que el ARNsf salival está presente en estructuras sensibles al detergente que lo protegen de la digestión por nucleasas.
Para comprobar si dichas estructuras sensibles al detergente eran complejos de ribonucleoproteína, realizaron tratamientos secuenciales con proteinasa K (PK) y ARNasa A/T1 en muestras de saliva. Para evitar que la PK digiriera las nucleasas, inhibieron la primera con fluoruro de fenilmetilsulfonilo (PMSF), disuelto en DMSO.
Tras ello, el ARNsf1 transcrito in vitro y añadido a la saliva no infectada, fue digerido por la ARNasa independientemente del tratamiento con PK (Fig. 2D). En la saliva infectada, observaron una digestión parcial del ARNsf por la ARNasa en ausencia de tratamiento con PK, lo que podría atribuirse al DMSO, utilizado para controlar el PMSF (Figs. 2B y 2E), que desestabiliza las membranas. A diferencia del ARNsf, el ARNg no fue tan sensible a la ARNasa independientemente del tratamiento (Fig. 2F), lo cual no concuerda con la protección por proteínas. Además, los datos sugieren que una fracción del ARNg se empaquetó en un compartimento resistente al DMSO, lo cual es previsible para los viriones del dengue, mientras que el ARNsf se empaquetó en un compartimento más sensible al DMSO.
El ARN antiinmune del dengue está presente en las vesículas extracelulares salivales
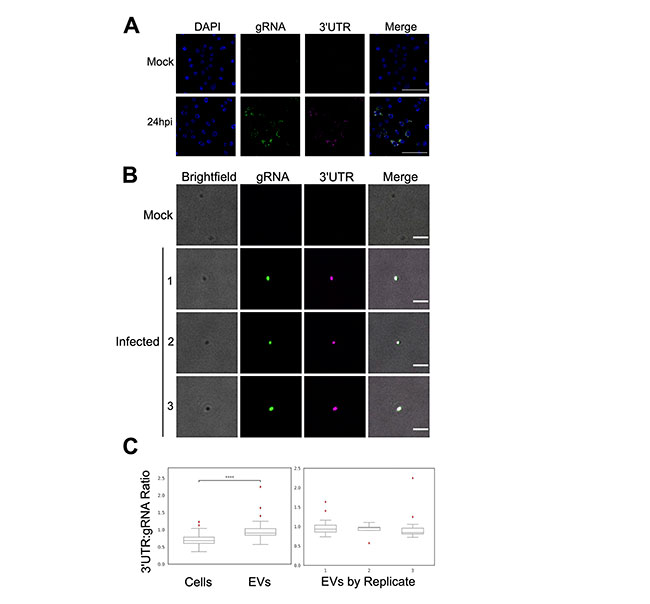
Dado que el ARN antiinmune del dengue se localiza en compartimentos sensibles a detergentes, postularon su probable presencia en las vesículas extracelulares (VE) salivales. Dada la escasez de reactivos disponibles para la identificación bioquímica de las VE de mosquitos, obtuvieron imágenes de la saliva de mosquitos mediante microscopía electrónica de transmisión (MET) para su identificación morfológica. La saliva de mosquitos, tanto infectada como no infectada, contenía estructuras esféricas rodeadas de membranas, similares a las VE observadas mediante MET en la saliva de garrapatas. Los diámetros de las partículas similares a VE observadas oscilaron entre ~100 y 800nm en la saliva no infectada y entre ~100 y 500nm en la saliva infectada. Así pues, concluyeron que la saliva de mosquitos, tanto infectada como no infectada con DENV2, contiene partículas similares a VE.
Por otra parte, utilizaron sondas de hibridación in situ con fluorescencia de ARN (RNA-FISH) de Oligopaints para determinar si los ARN virales en saliva infectada con DENV2 se encontraban dentro de partículas similares a EV. Tras esto, los nucleótidos 1-10.293 del genoma NGC de DENV2 fueron detectados por 240 oligos codificados como ARNg, mientras que los nucleótidos 10.294-10.717 fueron detectados por 7 oligos codificados como 3’UTR, que también detectan ARNsf.
Para comprobar si las sondas RNA-FISH detectaban específicamente los ARN virales, compararon la RNA-FISH de células C6/36 infectadas simuladas e infectadas con DENV2 y, como era de esperar, observaron señal solo en las células infectadas con DENV2 (Fig. 3A). Las células infectadas con DENV2 presentaron la tinción perinuclear canónica del ARN viral, con frecuente colocalización de las señales fluorescentes de ARNg y 3’UTR (Fig. 3A). Se obtuvo el mismo resultado al intercambiar los fluoróforos utilizados para detectar las sondas de ARNg y 3’UTR. La detección exclusiva de sondas de ARNg en etapas muy tempranas tras la infección en células C6/36 demostró la especificidad del marcaje, cuando se espera que el ARNsf sea indetectable.
Para evaluar aún más la especificidad de las sondas FISH, transfectaron un ARN 3’UTR PR6452 de DENV2 transcrito in vitro en células C6/36 dos horas antes del marcaje mediante ARN-FISH. Aunque existe una discordancia de 5 nucleótidos entre las secuencias 3’UTR de DENV2 NGC y PR6452, las imágenes de células C6/36 transfectadas mostraron marcaje con la 3’UTR, pero no con sondas de ARNg. Estos experimentos muestran que las sondas de ARN-FISH con código de barras de ARNg no detectaron secuencias virales 3’UTR y viceversa.
Dado que las sondas Oligopaints RNA-FISH demostraron ser específicas, las utilizaron para obtener imágenes de saliva de mosquitos no infectados e infectados con DENV2 (Fig. 3B). En saliva no infectada, observaron numerosas EV y, como era de esperar, la mayoría no se marcaron con las sondas Oligopaints (Fig. 3B). Y en saliva infectada con DENV2, observaron numerosas EV marcadas con sondas de ARNg y 3’UTR (Fig. 3B). Además, cabe decir que las EV positivas para 3’UTR resultaron ser de tamaño mediano/grande.
Las EV salivales infectadas con DENV2 están enriquecidas en el marcaje fluorescente 3’UTR en comparación con las células C6/36, ya que observaron una relación de fluorescencia 3’UTR:ARNg media de 0,685 (± 0,166 DE) en células C6/36 y 0,904 (± 0,294 DE) en las EV de saliva (Fig. 3C).
Para asegurar que la alta proporción de marcaje 3’UTR:ARNg observada en EVs no fuera un artefacto de los fluoróforos usados para visualizar oligos RNA-FISH hibridados, repitieron los experimentos RNA-FISH con oligos conjugados con fluoróforos secundarios intercambiados, y nuevamente observaron enriquecimiento de proporciones de marcaje 3’UTR:ARNg en EV salivales, que tuvieron una proporción de fluorescencia 3’UTR:ARNg media de 1.512 (± 0.766), en comparación con las células, que tuvieron una proporción de fluorescencia 3’UTR:ARNg media de 0.619 (± 0.513).
En general, los resultados Oligopaints RNA FISH demuestran que en muestras de saliva infectadas con DENV2, las estructuras similares a EV están enriquecidas en secuencias 3’UTR de DENV2. Este enriquecimiento es consistente con las altas proporciones ARNsf:ARNg detectadas mediante RT-qPCR anteriormente.
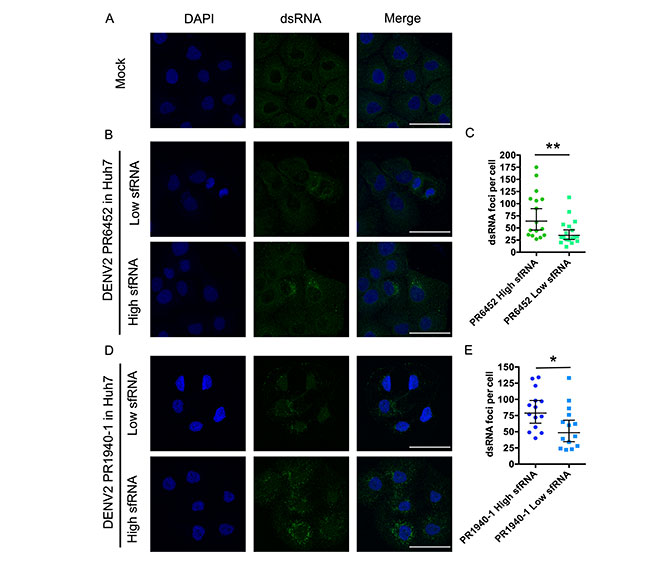
Los niveles elevados de ARN antiinmune del dengue se asocian con una mayor infectividad salival
Los científicos analizaron cómo los niveles de ARN antiinmune del dengue influyeron en las infecciones de células humanas mediadas por saliva. Para ello, aprovecharon sus observaciones de que los niveles de ARNsf salival variaban entre infecciones independientes de mosquitos.
Recolectaron muestras de saliva de mosquitos infectados con las cepas PR6452, PR1940 y PR9963 de DENV2 e identificaron aquellos con niveles similares de copias de ARNg, pero niveles de ARNsf sustancialmente diferentes y, por lo tanto, con proporciones ARNsf:ARNg bajas o altas. Dado que las proporciones ARNsf:ARNg varían significativamente entre mosquitos, trabajaron con muestras de saliva obtenidas de 40 mosquitos infectados con la misma cepa de DENV2, asumiendo que el impacto de cualquier saliva distinta se homogeneizaba entre las condiciones.
En cuatro experimentos independientes, uno con PR6452, dos con PR1940 y uno con PR9963, compararon la infección de células Huh-7 humanas con grupos de saliva con ratios ARNsf:ARNg bajos o altos. Para cada comparación entre grupos de saliva, infectaron células Huh-7 con el número equivalente de genomas virales en el mismo volumen total de saliva, lo cual se sabe que influye en la infectividad. Para ello, diluyeron muestras de saliva infectadas con saliva de mosquitos no infectados, como se describe en la siguiente Tabla 1.
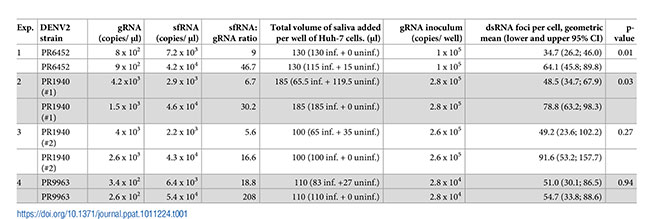
Como medida de la infección, cuantificaron los focos de replicación del genoma viral detectados por inmunofluorescencia con un anticuerpo anti-ARNdc. Validaron que el anticuerpo anti-ARNdc no detectó el ARNdc transfectado in vitro en células Huh-7. La media geométrica de focos de replicación por célula fue de 34,7 cuando las células se inocularon con saliva infectada con PR6452, con una proporción de ARNsf:ARNg inferior, frente a 64,1 para la saliva con una proporción mayor (Tabla 1 y Fig. 4B y 4C). Se obtuvieron resultados muy similares con los pools de saliva infectada con PR1940. Este efecto no se observó con los pools de saliva infectados con PR9963, posiblemente porque estas muestras de saliva tenían títulos virales de 5 a 10 veces menores que PR6452 y PR1940.
En estos experimentos, utilizaron el porcentaje de células Huh-7 infectadas como una segunda medida de infectividad y observaron que era mayor cuando las células se exponían a muestras de saliva infectadas con proporciones altas de ARNsf;ARNg.
Para determinar el impacto del ARN antiinmune del dengue salival en las células del sitio de la picadura, repitieron la infección salival con fibroblastos dérmicos humanos neonatales primarios (NHDF). Dado que la infección de los NHDF es mucho menos eficiente que la de las células Huh-7, cuantificaron el ARNg intracelular a las 72h postinfección. Si bien no detectaron genomas de DENV en células infectadas con proporciones bajas de ARNsf:ARNg, proporciones más altas resultaron en la detección de ARNg de DENV en NHDF, lo que sugiere que el ARNsf salival potencia la infección de las células humanas del sitio de la picadura. Con base en los resultados anteriores, propusieron que niveles altos de ARN antiinmune del dengue en la saliva de mosquitos potencian la infección por el virus del dengue en células humanas.
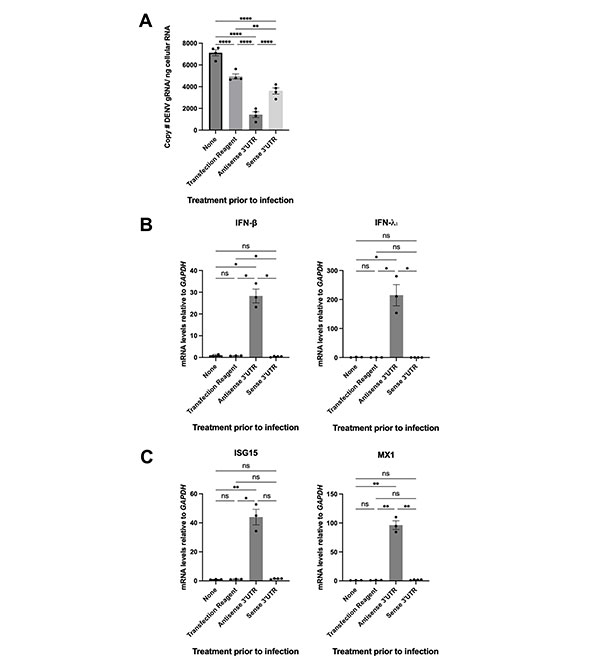
El ARN 3’UTR transcrito in vitro potencia la infectividad de DENV2 e inhibe la inducción y la señalización de interferón en células humanas
Para evaluar si el ARN antiinmune del dengue es responsable de las propiedades que favorecen la infección observada en la saliva, imitaron la administración de ARNsf transfectando un ARN 3’UTR de DENV2 5’-monofosforilado transcrito in vitro en células Huh-7 dos horas antes de la infección por DENV2.
Para controlar los efectos generales de la transfección de un ARN extraño sobre la infección, transfectaron un ARN 3’UTR antisentido 5’-monofosforilado. La transfección de cualquier ARN inhibió la infección, probablemente debido a la activación de las vías inmunitarias innatas y de estrés (Fig. 5). La transfección del ARN 3’UTR sentido aumentó significativamente los niveles de ARN genómico de DENV2 en las células infectadas (Fig. 5A). No se detectaron diferencias en la acumulación de ARN virales extracelulares en ninguna de las condiciones analizadas, lo cual no resulta sorprendente dados los cortos tiempos de infección. Estos datos, que coinciden con resultados publicados previamente, indican que la introducción de ARNf de DENV2 en células humanas aumenta la infectividad.
Para determinar si el aumento de la infección tras la transfección de la 3’UTR sentido se correlacionaba con la inhibición inmunitaria mediada por ARNsf, monitorizaron la inducción de IFN cuantificando la expresión de IFN-β e IFN-λ1 en las mismas muestras. Si bien la transfección de la 3’UTR antisentido aumentó los niveles de ARNm de IFN-β e IFN-λ1, la transfección de la 3’UTR sentido no lo hizo (Fig. 5B).
Además, monitorizaron la señalización de IFN cuantificando la expresión de los genes estimulados por IFN (ISG): ISG15 y resistencia al mixoma 1 (MX1). Como se observó, para los IFN la inducción de la expresión de ISG15 y MX1 por transfección de ARN fue reprimida notablemente por la 3’UTR sentido (Fig. 5C). En conjunto, estos datos respaldan la hipótesis de que la administración de ARN antiinmune del dengue mejora la infección por el virus del dengue al inhibir la inducción de IFN y la señalización posterior.
Discusión y Conclusión
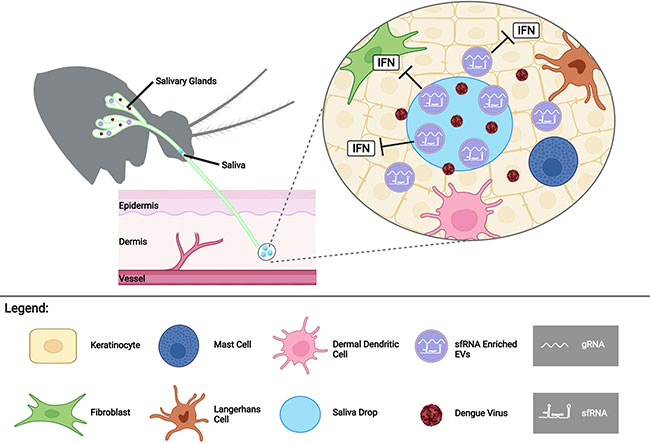
En conjunto, los resultados obtenidos en este trabajo indican que el ARN antiinmune del dengue potenciador de la infección se secreta en el lumen de las EV sensibles al detergente en la saliva del mosquito. Si bien es formalmente posible que el ARNsf esté empaquetado en viriones DENV2 en la saliva, como se propuso en un informe reciente que estudió las EV humanas, esto no es posible según la sensibilidad diferencial al DMSO observada para el ARNg y el ARNsf.
Las EV son vehículos bien conocidos de ARN codificantes y no codificantes, incluyendo ARN virales. Las observaciones de los científicos de las EV en la saliva del mosquito marcadas para DENV2 3’UTR y ARNg, son consistentes con estudios previos utilizando el virus del dengue y otros Flaviviridae. De hecho, las EV derivadas de células de mosquito infectadas con DENV se han aislado, caracterizado y se ha demostrado que contienen fragmentos de ARNg de dengue. Además, se ha sugerido que las EV derivadas de células de artrópodos en cultivo transmiten genomas virales a células HaCaT similares a queratinocitos humanos. Con base en estas consideraciones, proponen que el ARN antiinmune del dengue en la saliva está presente en las EV, que pueden entregar su carga a las células en la misma ubicación y, al mismo tiempo, que el virus se deposita en el sitio de la picadura, tal y como se muestra en la figura inferior.
Asimismo, se ha demostrado que el ARNsf tiene una potente función antiinmunitaria en células humanas, lo cual concuerda con los datos aquí presentados, que muestran que la transfección de un imitador de ARNsf previene la respuesta de IFN inducida por ARN. Con base en estos datos y según su observación de que niveles más altos de ARN antiinmune del dengue en saliva se correlacionan con una mayor infectividad del virus del dengue, postulan que el ARN antiinmune del dengue administrado a células cutáneas humanas durante la picadura, probablemente, debilite la respuesta inmunitaria innata local y otorgue al virus una ventaja que favorece la transmisión por mosquitos.
Además, el papel de las EV en la batalla entre patógenos y hospedadores no es inédito. En general, los hospedadores pueden transferir pequeños ARN para silenciar los genes de virulencia de los patógenos. En este informe, revelan otro nivel de complejidad y proponen que el virus del dengue aprovecha la biología de su vector invertebrado para aumentar la transmisión al hospedador humano.
Por último, cabe señalar que, a pesar de la presencia de algunas limitaciones en el trabajo debidas principalmente por los volúmenes tan pequeños con los que había que trabajar, sus métodos concluyeron la presencia de ARN antiinmune del dengue en las EV salivales y sugirieron firmemente que este ARNnc potenciaba la infectividad del virus del dengue (DENV2).
Fuente del artículo
Este artículo fue publicado originalmente en la revista PLoS Pathogens bajo la referencia: Yeh S-C, Strilets T, Tan W-L, Castillo D, Medkour H, Rey-Cadilhac F, et al. (2023) The antiimmune dengue subgenomic flaviviral RNA is present in vesicles in mosquito saliva and is associated with increased infectivity. PLoS Pathog 19(3): e1011224. https://doi.org/10.1371/journal.ppat.1011224; estando disponible con acceso libre en la propia web de PLoS Pathogens.
